Vermamoeba vermiformis
Vermamoeba vermiformis ist eine Art (Spezies) von Amöben (Amoebozoa) in der Gruppe Echinamoebida der Tubulinea.[1][2]
| Vermamoeba vermiformis | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
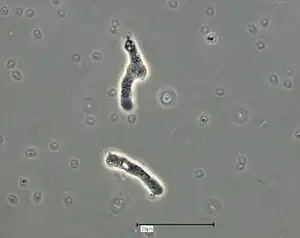
Trophozoiten von V. vermiformis, | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name der Gattung | ||||||||||||
| Vermamoeba | ||||||||||||
| Smirnov et al., 2011[1] | ||||||||||||
| Wissenschaftlicher Name der Art | ||||||||||||
| Vermamoeba vermiformis | ||||||||||||
| (F. C. Page, 1967) Smirnov et al., 2011[1][2][3] |

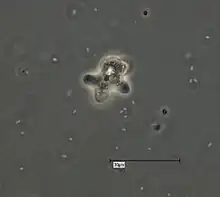




Die Gattung Vermamoeba ist monotypisch, d. h. sie hat nur die einzige beschriebene Art V. vermiformis. Allerdings führt die Taxonomie des National Center for Biotechnology Information (NCBI) noch einen Stamm als mögliche weitere Spezies mit der vorläufigen Bezeichnung Vermamoeba sp. SCN 42-11.[4]
Forschungsgeschichte
V. vermiformis wurde erstmals 1967 von F. C. Page als Hartmanella vermiformis beschrieben.[5] Da sich diese Spezies von den anderen Arten der Gattung Hartmanella deutlich unterscheidet, wurde sie im Jahr 2011 taxonomisch neu klassifiziert und in eine eigene neue Gattung Vermamoeba gestellt, wobei die Art ihren heutigen Namen bekam (Smirnov et al., 2011).[6] Der Unterschied zwischen den Gattungen Hartmanella und Vermaboeba besteht in der zylindrischen Form von Vermamoeba und dem Verhältnis von Länge und Breite.[7] Diese Umgruppierung wurde 2018 durch Delafont et al. per Genanalyse der 18S rRNA einer Reihe von Protisten, darunter V. vermiformis, bestätigt.[8][3]
Habitat
V. vermiformis gedeiht zwar auch im Boden, kommt aber häufiger im Wasser vor.[3][1] Darüber hinaus wurde diese Vermamöben in Schnee, Leitungswasser, Thermalwasser, Kühltürmen, Krankenhaus- und Haushaltsabwässern, Industriekompost und Bioaerosolen, bei Vögeln und Säugetieren einschließlich des Menschen nachgewiesen.[3]
Humanpathologie
Beim Menschen wurde V. vermiformis neben Arten der Gattung Acanthamoeba mit der Augenkrankheit Keratitis in Verbindung gebracht. Außerdem gibt es einen indirekten Bezug zur Legionärskrankheit, die das menschliche Atmungssystem betrifft. Vermamöben dienen als Wirt für pathogene, endosymbiotische Bakterien: außer den Legionella-Arten (Legionellen) sind das Stenotrophomonas maltophilia, Candidatus Rubidus massiliensis, Bacillus anthracis, Pseudomonas aeruginosa, Neochlamydia hartmannellae, Waddlia sowie andere Chlamydien-ähnliche Endosymbionten und Mycobacterium chelonae.[3][9]
Lebenszyklus
Im Lebenszyklus von V. vermiformis gibt es (wie bei Acanthamoeba) zwei Stadien, nämlich als Trophozoit (aktives Stadium) und als Zyste (Ruhestadium). Studien haben gezeigt, dass die Forma bei 4 °C kugelförmig und unbeweglich ist (Zyste), während der Trophozoit bei 50 °C lysiert wird (sich auflöst bzw. platzt). Die Zelllyse ist jedoch langsamer als die von Acanthamoeba.[3][9]
Trophozoit
Der Trophozoit hat eine länglich-zylindrisch Form. Diese ist nicht starr, denn der Einzeller kann sich mit Hilfe von Pseudopodien (Scheinfüßchen) fortbewegen. Diese ermöglichen es auch der Amöbe, sich zu ernähren und zu vermehren. Die Zellen sind einkernig mit einem zentralen dichten und homogenen Karyoplasma (Kernplasma). Mehrere Mitochondrien mit röhrenförmigen Cristae sind über das Zytoplasma verteilt.[3][9]
Die Trophozoiten erscheinen als aus monopodialen, langen, wurm- oder nacktschneckenartigen Zellen mit einem körnig erscheinenden Zytoplasma und einer leicht hyalinen (transparenten) vorderen Zone. Die Form wird durch Temperatur, pH-Wert und osmotischen Druck beeinflusst. Wenn sie ihre Richtung ändern, können vorübergehend zwei oder mehr Scheinfüßchen entstehen (bi- oder multipodiale Form).[3][9]
Zyste
Die Zyste hat eine kugelförmige Form mit einer zweischichtigen Zellwand ohne Poren (Ostiolen), die Proteine und eine geringe Menge an Glukose-Polymeren (aber keine Zellulose) enthält. Die Zystenbildung erfolgt in lebensfeindlichen Umgebungen, die beispielsweise nährstoffarm sind oder einen hohen osmotischem Druck erzeugen. Der Umwandlung zu Zyste (englisch encystment) dauert 9 Stunden, wobei sich in der 6. bis 9. Stunde die zweilagige Zellwand bildet. Die doppelte Zellwand und ihre Zusammensetzung sind für die kugelige Form der Zysten und ihre Widerstandsfähigkeit unter Nährstoffmangel, Hitze, Kälte, Austrocknung und Bioziden verantwortlich.[3][9]
Systematik
Die hier angegebene Systematik folgt in erster Linie dem World Register of Marine Species (WoRMS),[1] in zweiter der Taxonomie des National Center for Biotechnology Information (NCBI):
- (Phylum) Amoebozoa
Bis heute wurden viele Vermamoeba-Stämme aus verschiedenen Teilen der Welt charakterisiert, z. B. aus iranischen Mineralquellen, Myanmar, Spanien (Kanarische Inseln), Mittel- und Norditalien, Frankreich, Südkorea, den USA, Pakistan, England und China.[3] Die Spezies ist also ein Kosmopolit.
Genom
Es wurde bisher (Stand 2022) nur ein Entwurf einer Genomsequenz veröffentlicht, nämlich die des Stammes CDC-19.[3] Dessen Genom-Sequenz hat eine Gesamtlänge von 59.550.895 bp (Basenpaaren) und kodiert 22.483 Gene bei einem G+C-Gehalt von 41,7 %. Die Analysen ergaben eine geschätzte Anzahl von 7.220 ORFans, d. h. mutmaßliche Offene Leserahmen (ORFs) ohne Übereinstimmung in öffentlichen Gendatenbanken, sowie 2.829 Gene mit unbekannter Funktion. Die phylogenomische Analyse ergab, dass unter diesen zehn die beste homologe Übereinstimmung in der bakteriellen Gruppe Candidate Phyla Radiation (CPR-Gruppe) hatten. Darüber hinaus wiesen 185 Gene die besten Übereinstimmungen mit Viren des Phylums Nucleocytoviricota (NCLDV), mit den weitreichendsten Beziehungen zu Klosneuvirinae wie den Gattungen „Klosneuvirus“ und Theiavirus (mit dem Bodo-saltans-Virus). Knapp 60 % der Gene stimmten mit anderen Eukaryoten überein (knapp 20 % davon Amöben), gut 10 % mit Bakterien, 0,6 % mit Archaeen und 0,8 % mit Viren, darunter zwei Gene mit dem Ralstonia-Phagen phiRSL1 (Mieseafarmvirus RSL1)[11] und ein Gen mit dem Synechococcus-Phagen S-SKS1 (Llyrvirus SSKS1),[12] beide aus der Klasse Caudoviricetes vom Morphotyp der Myoviren. Es wurden 54 Gene identifiziert, die mit Abwehrmechanismen in Verbindung stehen; im Schnitt gibt es 3,5 Introns pro Gen.[3]
Viren
Die V. vermiformis parasitierenden Viren umfassen nach Geballa-Koukoulas et al. (2022) zu diesem Zeitpunkt 26 beschriebene Isolate, die acht verschiedenen Taxa (Virenarten) an gehören, sieben davon lytisch, eines nicht. Entsprechend der Größe ihrer Viruspartikel (Virionen), der Replikaionszteit und der Zahl der kodierten tRNAs (Transfer-RNAs) gliederten sich diese in drei verschiedene Gruppen. Neue Co-Kultivierungstechniken haben die Untersuchung von Vermamoeba-Viren seit etwa 2016 stark vereinfacht.[3]
„Faustovirus“
V. vermiformis kann als Wirt von acht Stämmen der vorgeschlagenen Gattung „Faustovirus“ dienen, die mit dem Afrikanischen Schweinepest-Virus (ASFV, Asfarviridae) verwandt ist.[3]
„Kaumoebavirus“
_Mature.png.webp)
Auch die Gattung „Kaumoebavirus“ ist derzeit (Stand Mitte Mai 2023) noch nicht vom International Committee on Taxonomy of Viruses (ICTV) bestätigt. In phylogenetischen Untersuchungen gruppiert sie zwischen ASFV und „Faustovirus“, was darauf hindeutet, dass sie ein Mitglied der Familie der Asfarviridae ist. Diese phylogenetische Position spiegelt sich in gemeinsamen Merkmalen zwischen diesen Viren wider. Insbesondere teilen „Faustovirus“ und „Kaumoebavirus“ die gleiche Morphologie: ein ikosaedrisches Kapsid mit einem Durchmesser von etwa 200 nm, die Dauer des Replikationszyklus und ein gespleißtes Hauptkapsidprotein-Gen, das für ein Protein etwa gleicher Größe kodiert.[3]
„Orpheovirus“
Die Viruspartikel von „Orpheovirus“ sind eiförmig mit einer Pore (Ostiole) am Ende, durch die die DNA auf die Wirtszelle übertragen wird. Die Partikelstruktur teilt einige morphologische Eigenschaften mit Cedratviren, Pithoviren und Pandoraviren. Darüber hinaus zeigen sowohl phylogenetische und andere Analysen, dass Cedratviren und Pithoviren (nicht jedoch Pandoraviren) eng mit „Orpheovirus“ verwandt sind. Die Genomgröße ist mit 1,4 Mbp (Mega-Basenpaare) riesig verglichen mit „Faustovirus“ und „Kaumoebavirus“. Das Genom ist offenbar ringförmig. Das Virus gelangt durch den Prozess der Phagozytose in die Vermamoeba-Zelle.[3]
Tupanvirus
Die im April 2023 vom ICTV bestätigte Gattung Tupanvirus besteht aus zwei Spezies von Isolaten aus einem brasilianischen Natronsee im Pantanal und Tiefsee-Bodensedimenten aus 3.000 m Tiefe (bei Campos dos Goytacazes).[13][14] Beide Arten verfügen über ein lineares Genom. Sie können sich sowohl in Vermamoeba und Acanthamoeba, als auch in anderen Amöben wie Willaertia magna und dem Schleimpilz Dictyostelium discoideum vermehren. Die Virionen der Tupanviren haben eine ungewöhnliche Struktur, die durch ein halb-ikosaedrischen, Mimivirus-ähnliches Kapsid gekennzeichnet ist, an dem ein länglicher zylindrischer „Schwanz“ befestigt ist[3] (der sich aber von den drei Caudoviricetes-Morphotypen unterscheidet). Dieser kann unterschiedlich groß sein, so dass die Gesamtlänge im Extremfall 2 μm überschreiten kann.[3]
„Clandestinovirus“

Das erste nichtlytische Isolat eines Vermamoeba-Virus (in der vorgeschlagenen Gattung „Clandestinovirus“, CLV), wurde 2021 beschrieben (Rolland et al., 2021). Dieses Virus wurde aus einer gemischten Co-Kultur abgetrennt, die mit einem anderen Riesenvirus, „Faustovirus“ ST1, aus einer Umweltprobe assoziiert war und trägt ebenfalls die Bezeichnung ST1 (CLV-ST1).[3]
Yasminevirus
Die ebenfalls im April 2023 vom ICTV bestätigte Gattung Yasminevirus mit der Art Y. saudimassiliense wurde wie „Kaumoebavirus“ aus Abwasser in Dschidda, Saudi-Arabien, isoliert (Stamm GU-2018). Das Virus ähnelt dem Bodo-saltans-Virus (BsV, Theiavirus salishense) und wurde vom ICTV daher in dieselbe Unterfamilie Klosneuvirinae gestellt. Es wird angenommen, dass sein Virionenkern ist von dünnen Membranen umgeben ist, die vom ikosaedrischen Kapsid mit Durchmesser 330 nm umgeben sind.[13][14][3]
Fadolivirus
Die ebenfalls im April 2023 vom ICTV bestätigte Gattung Fadolivirus wurde mit Stamm FV1/VV64 im Jahr 2020 aus Abwasser in Sidi bel Abbès in Algerien gefunden. Es handelt sich um ein lytisches Virus, sein Kapsid hat eine ikosaedrischen Struktur mit einem Durchmesser von ca. 300 nm; von Kapsid gehen kurze Fibrillen aus. Die Gattung gehört ebenfalls zur Unterfamilie Klosneuvirinae.[13][14][3]
Etymologie
Das (ältere) Artepitheton vermiformis ist lateinisch und bedeutet „wurmförmig“, was auf die Form der Trophozoiten hinweist. Der (neuere) Gattungsname Vermamoeba bedeutet „Wurmamöbe“ und wiederholt diesen Bezug.
Weblinks
- Vermamoeba vermiformis, auf OneZoom
- Vermamoeba, auf Lifemap, NCBI Version
- Vermamoeba vermiformis (F.C.Page, 1967), Hartmannella vermiformis Page, 1967, auf GBIF.
- Hartmannella vermiformis Page, 1967, auf ITIS
- Alexey V. Smirnov, Ema Chao, Elena S. Nassonova, Thomas Cavalier-Smith: A Revised Classification of Naked Lobose Amoebae (Amoebozoa: Lobosa). In: Protist, Band 162, Nr. 4, Oktober 2011, S. 545-570; doi:10.1016/j.protis.2011.04.004, ResearchGate, PMID 21798804, EuropePMC, Epub 28. Juli 2011. Siehe insbes. Fig. 8 D–E: Vermamoeba (=Hartmannella) vermiformis, Stamm Valamo; F–G: weitere Stämme.
Einzelnachweise
-
WoRMS: Vermamoeba Smirnov et al., 2011 (Genus). Environment: fresh.
Anm.: Bitte Schieber 'marine only' und ggf. 'extant only' aus 'off' setzen. - NCBI Taxonomy Browser: Vermamoeba vermiformis, homotypic synonym: Hartmannella vermiformis Page, 1967 (species), Nucleotide: txid5778[Organism:noexp] Vermamoeba vermiformis.
- Khalil Geballa-Koukoulas, Bernard La Scola, Guillaume Blanc, Julien Andreani: Diversity of Giant Viruses Infecting Vermamoeba vermiformis. In: Frontiers in Microbiology, Band 13, 22. April 2022, S. 808499; doi:10.3389/fmicb.2022.808499 , PMID 35602053, PMC 9116030 (freier Volltext), PDF.
- NCBI Taxonomy Browser: Vermamoeba, Details: Vermamoeba (genus).
- Frederick C. Page: Taxonomic Criteria for Limax Amoebae, with Descriptions of 3 New Species of Hartmannella and 3 of Vahlkampfia. In: The Journal of Protozoology, Band 14, August 1967, S. 499–521; doi:10.1111/j.1550-7408.1967.tb02036.x.
- Alexey V. Smirnov, Ema Chao, Elena S. Nassonova, Thomas Cavalier-Smith: A Revised Classification of Naked Lobose Amoebae (Amoebozoa: Lobosa). In: Protist, Band 162, Nr. 4, Oktober 2011, S. 545-570; doi:10.1016/j.protis.2011.04.004, ResearchGate, PMID 21798804, EuropePMC, Epub 28. Juli 2011. Siehe insbes. Fig. 8 D-E: Vermamoeba (=Hartmannella) vermiformis, Stamm Valamo; F-G: weitere Stämme.
- Jong Soo Park: First Record of Potentially Pathogenic Amoeba Vermamoeba vermiformis (Lobosea: Gymnamoebia) Isolated from a Freshwater of Dokdo Island in the East Sea, Korea. In: Animal Systematics, Evolution and Diversity, Band 32, Nr. 1, 31. Januar 2016, S. 1–8; doi:10.5635/ASED.2016.32.1.001.
- Vincent Delafont, Marie-Helene Rodier, Elodie Maisonneuve, Estelle Cateau: Vermamoeba vermiformis: a Free-Living Amoeba of Interest. In: Environmental Microbiology, Band 76, November 2018, S. 991–1001; doi:10.1007/s00248-018-1199-8, PMID 29737382, Epub 8. Mai 2018.
- Patrick Scheid: Vermamoeba vermiformis - A Free-Living Amoeba with Public Health and Environmental Health Significance. In: The Open Parasitology Journal, Band 7, Nr. 1, April 2019, S. 40-47; doi:10.2174/1874421401907010040, ResearchGate.
-
WoRMS: Hartmannella vermiformis Page, 1967 (Species). Environment: fresh.
Anm.: Bitte Schieber 'marine only' und ggf. 'extant only' aus 'off' setzen.
Der Eintrag berücksichtigt noch nicht die Reklassifizierung durch Smirnov et al. (2011). - NCBI Taxonomy Browser: Ralstonia phage phiRSL1, equivalent: Ralstonia phage RSL1, … (no rnk).
- NCBI Taxonomy Browser: Synechococcus phage S-SKS1, equivalent: Cyanophage S-SKS1 (no rank).
- ICTV: Master Species Lists § ICTV Master Species List 2022 MSL38 v1 (xlsx), 8. April 2023.
- Frank O. Aylward, Jônatas S. Abrahão, Corina P. D. Brussaard C, Matthias G. Fischer, Mohammad Moniruzzaman, Hiroyuki Ogata, Curtis A. Suttle: Create 3 new families, 3 subfamilies, 13 genera, and 20 new species within the order Imitervirales (phylum Nucleocytoviricota) and rename two existing species (zip:docx). Vorschlag 2022.004F an das ICTV vom Oktober 2021.
