Purinbiosynthese
Die Purinbiosynthese (genauer: IMP-de novo-Synthese) ist der Stoffwechselweg, der alle Lebewesen dazu befähigt, Purine aus einfachen Ausgangsstoffen herzustellen. Purine haben eine überragende Bedeutung als Bestandteil der Erbinformation, als Energieträger (ATP) und als Basis für die Synthese weiterer wichtiger Stoffe.

| Übergeordnet |
| Synthese von IMP |
| Gene Ontology |
|---|
| QuickGO |
Die Purine werden im Organismus nicht als freie Moleküle synthetisiert, sondern stets als Nukleotide. Ausgangspunkt der Purinsynthese ist das α-D-Ribose-5-phosphat, ein Zwischenprodukt des Pentosephosphatzyklus. Das Endprodukt nach elf Schritten ist das Inosinmonophosphat (IMP), das Nukleotid des Hypoxanthins, welches in weiteren Schritten zu den Nukleotiden des Xanthinosins, Adenosins oder Guanosins umgebaut wird.
Die Purinsynthese findet beim Menschen in allen Zelltypen statt, die einen aktiven Zellkern haben.
Übersicht
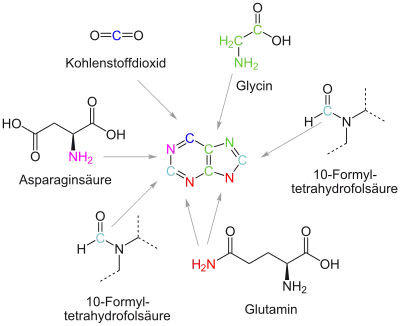
Das Grundgerüst des Purins wird schrittweise aufgebaut, wobei verschiedene Moleküle die einzelnen Bestandteile liefern:
Genomische Organisation in verschiedenen Spezies
Der Ablauf und die Gene der de novo-Synthese sind hochkonserviert.[1] Obwohl die de novo-Synthese quasi ubiquitär ist, gibt es zwischen den Arten doch Unterschiede in der Organisation der Enzyme und der dafür codierenden Gene. Die Zahl der Gene, welche an den zehn enzymatischen Schritten beteiligt sind, nimmt von Prokaryoten zu Eukaryoten ab, jedoch nimmt die Komplexität der Enzyme in gleicher Weise zu. Es bilden sich sogenannte Clustergene, die für multifunktionelle Enzyme codieren.
Beim Menschen sind die katalytischen Domänen der Einzelschritte (2,5,3), (6,7) und (9,10) jeweils auf einem Protein zu finden (PRPP-Synthese zählt als Schritt Null).
Einzelschritte und Zwischenprodukte
PRPP
 + ATP
+ ATP  + AMP
+ AMP
α-D-Ribose-5-phosphat wird mittels der Ribosephosphat-Diphosphokinase zu α-D-5-Phosphoribosyl-1-pyrophosphat (PRPP) umgesetzt.
PRA
 +
+  + H2O
+ H2O  +
+ ![]() + PPi
+ PPi
Aus PRPP und Glutamin entsteht 5-Phosphoribosylamin (PRA) und Glutamat, mithilfe der Amidophosphoribosyltransferase. Dieser Reaktionsschritt ist festlegend, das heißt, aus den Produkten dieser und der nächsten Reaktionen kann von nun an nur noch IMP hergestellt werden.
Die im Gleichgewicht befindliche Reaktion wird durch Hydrolyse des Diphosphats stark nach rechts verschoben. Als Hemmstoffe fungieren IMP, GMP und AMP.[2]
GAR
 +
+ ![]() + ATP
+ ATP  + ADP + Pi
+ ADP + Pi
Die Phosphoribosylamin-Glycin-Ligase-Domäne des trifunktionellen Purinsyntheseproteins erleichtert die Addition von Glycin an 5-Phosphoribosylamin (PRA) zu Glycinamidribonukleotid (GAR).
FGAR
 + 10-Formyl-THF
+ 10-Formyl-THF 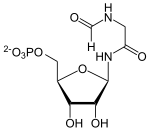 + THF
+ THF
Die Phosphoribosylglycinamid-Formyltransferase-Domäne (GART) des trifunktionellen Purinsyntheseproteins katalysiert die Formylierung von Glycinamidribonukleotid (GAR) zu Formylglycinamidribonukleotid (FGAR) mittels Formyltetrahydrofolat.
FGAM
 +
+  + ATP + H2O
+ ATP + H2O  +
+ ![]() + ADP + Pi
+ ADP + Pi
Phosphoribosylformylglycinamid (FGAR) und Glutamin werden zu Phosphoribosylformylglycinamidin (FGAM) und Glutaminsäure umgesetzt. Katalysierendes Enzym ist die FGAM-Synthase.
AIR
 + ATP
+ ATP 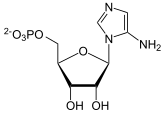 + ADP + Pi + H2O
+ ADP + Pi + H2O
Die Phosphoribosylformylglycinamidin-Cyclo-Ligase-Domäne des trifunktionellen Purinsyntheseproteins cyclisiert Formylglycinamidinribonukleotid (FGAM) zu 5-Aminoimidazolribonukleotid (AIR).
CAIR
 + CO2
+ CO2 
5-Aminoimidazolribonukleotid (AIR) wird mittels der Phosphoribosylaminoimidazol-Carboxylase-Domäne (AIRC) von ADE2 zu 5-Aminoimidazol-4-carboxylatribonucleotid (CAIR) carboxyliert.
SAICAR
 +
+ ![]() + ATP
+ ATP  + ADP + Pi
+ ADP + Pi
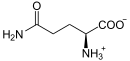
CAIR und Asparaginsäure werden ligiert, mit Verbrauch von ATP, katalysiert von der SAICAR-Synthase-Domäne von ADE2.
FAICAR
 + 10-Formyl-THF
+ 10-Formyl-THF 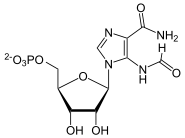 + THF
+ THF
AICAR wird zu FAICAR formyliert. Katalysator ist die AICAR-Formyltransferase-Domäne des bifunktionellen Purinsyntheseproteins.
IMP

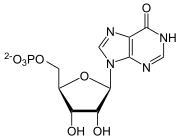 + H2O
+ H2O
FAICAR cyclisiert unter Wasserabspaltung zu IMP mittels der IMP-Cyclohydrolase-Domäne des bifunktionellen Purinsyntheseproteins.
Pathologie
Beim Menschen sind mehrere Mutationen in Genen bekannt, die für Enzyme der Purinsynthese codieren und zu seltenen erblichen Stoffwechselkrankheiten führen können:
Mutationen im PRPS1-Gen der Ribosephosphat-Diphosphokinase können zu einer Überaktivität des Enzyms, und diese zu erhöhtem erblichem Risiko für Gicht führen. Andere PRPS1-Mutationen verringern die Enzymaktivität und sind die Ursache für das so genannte Rosenberg-Chutorian-Syndrom und eine Form der Gehörlosigkeit (ARTS).[3][4]
Mutationen im ADSL-Gen der Adenylosuccinat-Lyase sind für Adenylosuccinat-Lyase-Mangel verantwortlich, eine seltene Erbkrankheit.[5]
Mutationen im ATIC-Gen des bifunktionellen Purinsyntheseproteins können die sehr seltene schwere AICA-Ribosidurie verursachen.[6]
Einzelnachweise
- Wagner, K. G. & Backer, A. I. (1992). Dynamics of nucleotides in plants studied on a cellular basis. Int. Rev. Cytol. 134, 184
- Jassal/reactome: 5-phospho-alpha-D-ribose 1-diphosphate (PRPP) + H2O + L-glutamine <=> 5-phosphoribosylamine + L-glutamate + pyrophosphate
- UniProt P60891
- Letale Ataxie mit Schwerhörigkeit und Optikusatrophie. In: Orphanet (Datenbank für seltene Krankheiten).
- orphaNet: Adenylosuccinat-Lyase-Mangel
- AICA-Ribosidurie. In: Orphanet (Datenbank für seltene Krankheiten).