Picornaviridae
Die Familie der Picornaviren (Picornaviridae) umfasst unbehüllte Viren mit einer einzelsträngigen, linearen RNA mit positiver Polarität als Genom. Die Viren dieser Familie gehören mit einer Größe von 22 bis 30 nm zu den kleinsten Viren, was zur Namensgebung pico (lat. für sehr klein) und rna für das Genom führte.[2]
| Picornaviridae | ||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

| ||||||||||||||
| Systematik | ||||||||||||||
| ||||||||||||||
| Taxonomische Merkmale | ||||||||||||||
| ||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||
| Picornaviridae | ||||||||||||||
| Links | ||||||||||||||
|
Picornaviren kommen bei einer Vielzahl von Wirbeltieren vor und verursachen sehr unterschiedliche Erkrankungen, z. B. eine harmlose Erkältung, Durchfallerkrankungen, Schleimhautentzündungen oder Infektionen des Zentralnervensystems. Die zahlreichen Arten der Picornaviren werden typischerweise in viele Subtypen unterteilt, da sie sich durch eine große Oberflächenvarianz und die damit einhergehende antigenetische Variabilität auszeichnen; bislang wurden ca. 370 Typen klassifiziert. Wichtige Vertreter der Picornaviridae sind beispielsweise beim Menschen das Hepatitis-A-Virus, in der Gattung Enterovirus das Poliovirus, die Rhinoviren (häufigste Erreger von Erkältungskrankheiten) und die Coxsackie-Viren, bei Tieren das Maul-und-Klauenseuche-Virus.
Morphologie

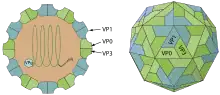
Die Virionen (Viruspartikel) der Picornaviridae haben eine runde Gestalt und sind etwa 22–30 nm im Durchmesser groß. Sie bestehen aus einem unbehüllten, ikosaedrischen Kapsid, das aus vier Virusproteinen VP1, VP2, VP3 und VP4 aufgebaut ist. Bei einigen Virusspezies ist noch ein Vorläuferprotein VP0 in geringen Mengen im Kapsid enthalten, aus dem während der Reifung der Partikel durch proteolytische Spaltung die Proteine VP2 und VP4 entstehen. Die vier Strukturproteine VP1-4 bilden zusammen ein Kapsomer, bei dem das VP4 die innere Kapsidseite auskleidet und über seine positiv geladenen Aminosäurereste mit der viralen RNA assoziiert ist. In einem Picornavirus-Kapsid lagern sich 60 Kapsomere zu einem Ikosaeder (T=1) zusammen. Die Oberfläche des Virions wird nur von den drei Proteinen VP1-3 gebildet, so dass nur diese für die antigenetischen Eigenschaften und die Einteilung in Serotypen verantwortlich sind.
Die Picornaviren sind aufgrund der Abwesenheit einer Virushülle sehr stabil gegenüber Alkoholen (Ethanol, 2-Propanol) und milden Detergenzien (Seife). Die Spezies der Gattungen Enterovirus und Hepatovirus sind darüber hinaus auch in Gegenwart starker Detergentien und längere Zeit bei pH-Werten unter 3,0 stabil, was ihnen eine außergewöhnliche Umweltresistenz verleiht. Aufgrund dieser Säurestabilität werden die Viren dieser beiden Gattungen auch durch das saure Milieu im Magen nicht inaktiviert;[2] daher geht der Infektionsweg dieser Viren überwiegend über den Verdauungstrakt, von dem aus sie auch weitere Zielorgane (ZNS, Lunge) erreichen können. Dafür sind Enteroviren empfindlich gegenüber Austrocknen sowie mäßigem Erhitzen (50 °C).[2] Alle anderen säurelabilen Picornaviren infizieren durch Tröpfchen- und Schmierinfektion bevorzugt den Nasen-Rachen-Raum. Rhinoviren sind empfindlicher; sie sind nur bei einem pH-Wert von 6,0-7,5 stabil und äußerst temperaturempfindlich.[2]
Genom
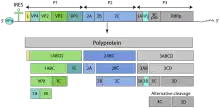
Das virale Genom besteht aus einer einzelsträngigen RNA mit positiver Polarität. Die Länge der RNA variiert zwischen den Gattungen von 7,2 (Rhinoviren) bis 8,5 (Aphthovirus) kB. Zwischen zwei nichtcodierenden Bereichen am 3'- und 5'-Ende liegt ein einziger offener Leserahmen (ORF) für ein virales Vorläufer-Polyprotein, das noch während der Translation in einzelne Virusproteine gespalten wird. Am 3'-Ende befindet sich ein für positivsträngige RNA-Viren typischer poly-A-Schwanz. Der RNA-Abschnitt am 5'-Ende vor dem Startcodon ist durch zahlreiche Basenpaarungen innerhalb des RNA-Moleküls zu einer komplexen Sekundärstruktur gefaltet, die funktionell die Aktivität einer IRES (internal ribosomal entry site) zeigt. Diese Struktur dient der Initiation der Translation an den Ribosomen und wurde erstmals bei Picornaviren beschrieben.
Systematik
Die folgende Systematik entspricht dem ICTV-Stand vom März 2020,[1][3] ergänzt um die vorgeschlagene Einteilung in sog. Supergruppen (im Rang von Unterfamilien) und weitere vorgeschlagene Gattungen (in Anführungszeichen).[4] Es ist nur eine Auswahl der Spezies angegeben. Im März 2021 hat das ICTV die Supergruppen als Unterfamilien anerkannt.[5]
- Familie Picornaviridae
- Unterfamilie Caphthovirinae (ehemals „Supergruppe 1“)
- Gattung Ailurivirus
- Gattung Aphthovirus
- Gattung Bopivirus
- Gattung Cardiovirus[6][7]
- Spezies Cardiovirus A (mit Encephalomyocarditis virus = ECM-Virus, Mengovirus)
- Spezies Cardiovirus B (mit Theiler's murine encephalomyeltits virus = TMEV, Theilovirus)
- Spezies Cardiovirus C (mit Boone Cardiovirus = BCV)
- Gattung Cosavirus[8]
- Gattung Erbovirus
- Gattung Hunnivirus
- Gattung Malagasivirus
- Gattung Mischivirus
- Gattung Mosavirus
- Gattung Mupivirus
- Gattung Senecavirus
- Gattung Teschovirus
- Gattung Torchivirus
- Gattung Tottorivirus
- Unterfamilie Kodimesavirinae (ehemals „Supergruppe 2“)
- Gattung Danipivirus
- Gattung Dicipivirus
- Gattung Gallivirus[9]
- Spezies Gallivirus A (mit Truthahn-Gallivirus alias Turkey gallivirus)
- Spezies „Hühner-Gallivirus 1“ alias „Chicken gallivirus 1“[10]
- Spezies „Gallivirus Pf-CHK1/GV“
- Spezies „Red-necked stint gallivirus“
- Gattung Hemipivirus
- Gattung Kobuvirus[11][8]
- Spezies Aichivirus A
- Spezies Aichivirus B
- Spezies Aichivirus C
- Spezies Aichivirus D
- Spezies Aichivirus E
- Spezies Aichivirus F
- Gattung Livupivirus
- Gattung Ludopivirus
 Virion der Gattung MegrivirusGattung Megrivirus[12] (Stand: 28. September 2023)
Virion der Gattung MegrivirusGattung Megrivirus[12] (Stand: 28. September 2023) Genom, Gattung Megrivirus
Genom, Gattung Megrivirus- Spezies Megrivirus A (mit Gänse-Megrivirus alias Goose megrivirus, Megrivirus A2, Megrivirus A3, Megrivirus B3CP-APO)
- Spezies Megrivirus B (mit Picornavirus HK21 alias Pigeon mesivirus 1 und Pigeon mesivirus 2)
- Spezies Megrivirus C (mit Hühner-Megrivirus alias Chicken megrivirus, Megrivirus A1CP-CPOL, Megrivirus C2, Megrivirus C3, Turkey hepatitis virus)
- Spezies Megrivirus D (mit Harrier picornavirus 1)
- Spezies Megrivirus E (mit Pinguin-Megrivirus alias Penguin megrivirus)[13]
- Spezies „Truthahn-Hepatitis-Virus“ (syn. „Melegrivirus A“, mit Turkey hepatitis virus 0091.1, Turkey hepatitis virus 2993D)
- Spezies „Megrivirus der Eigentlichen Säbelschnäbler“ („Avocet megrivirus“)
- Spezies „Kastanienenten-Megrivirus“ („Chestnut teal megrivirus“)
- Spezies „Enten-Megrivirus“ („Duck megrivirus“)
- Spezies „Augenbrauenenten-Megrivirus“ („Pacific black duck megrivirus“)
- Spezies „Rosenohrenten-Megrivirus“ („Pink-eared duck megrivirus“)
- Spezies „Rotkopf-Regenpfeifer-Megrivirus“ („Red-capped plover megrivirus“)
- Spezies „Truthahn-Megrivirus“ („Turkey megrivirus“)
- Spezies „Brautenten-Megrivirus“ („Wood duck megrivirus“)
- Gattung Myrropivirus
- Gattung Oscivirus,[14]
- Spezies Oscivirus A (mit Oscivirus A1, früher „Turdivirus 2“; Oscivirus A2, früher „Turdivirus 3“[10])
- Gattung Passerivirus[15]
- Spezies Passerivirus A (mit Passerivirus A1, früher „Turdivirus 1“[10])
- Gattung Pemapivirus
- Gattung Poecivirus
- Gattung Pygoscepivirus, früher „Pinguvirus“[10]
- Spezies Pygoscepivirus A, früher „Pingu picornavirus“ (gefunden bei Eselspinguinen (Pygoscelis papua))[10]
- Gattung Rafivirus[16]
- Spezies Rafivirus A (mit Tortoise Rafivirus A)[10]
- Spezies Rafivirus B
- Spezies Rafivirus C (mit Hainan gekko similignum picornavirus)
- Gattung Rajidapivirus
- Gattung Rosavirus[17]
- Spezies Rosavirus A (mit Rosavirus A1 mit Rosavirus M-7; Rosavirus A2)
- Spezies Rosavirus B (mit Norway rat rosavirus)
- Spezies Rosavirus C
- Gattung Sakobuvirus[18]
- Spezies Sakobuvirus A (mit Feline sakobuvirus A)
- Spezies Salivirus A (mit Human klassevirus 1, Salivirus A SZ1, Salivirus CH, 1, Salivirus NG-F1, Salivirus NG-J1, Salivirus SH1)
- Gattung Sicinivirus[20]
- Spezies Sicinivirus A (mit Sicinivirus Pf-CHK1/SiV)
- Gattung Symapivirus
- Gattung Tropivirus
- Spezies Tropivirus A
- Spezies Tropivirus B
- Unterfamilie Ensavirinae (ehemals „Supergruppe 3“)
- Gattung Anativirus
- Gattung Boosepivirus
- Gattung Diresapivirus
- Gattung Enterovirus (inklusive ehemalige Gattung „Rhinovirus“)
- Gattung Felipivirus
- Gattung Parabovirus
- Gattung Rabovirus
- Gattung Sapelovirus
- Unterfamilie Paavivirinae (ehemals „Supergruppe 4“)
- Gattung Aalivirus
- Gattung Aquamavirus
- Gattung Avihepatovirus
- Spezies Enten-Hepatitis-Virus 1 (DHV-1)
- Spezies Enten-Hepatitis-Virus 3 (DHV-3)
- Gattung Avisivirus
- Gattung Crohivirus
- Gattung Grusopivirus
- Gattung Kunsagivirus
- Gattung Limnipivirus
- Gattung Orivirus
- Gattung Parechovirus[8]
- Spezies Parechovirus A
- Spezies Parechovirus B (mit Ljungan virus)
- Spezies Parechovirus C
- Spezies Parechovirus D
- Spezies Parechovirus E
- Spezies Parechovirus F
- Gattung Pasivirus
- Gattung Potamipivirus
- Gattung Shanbavirus
- Unterfamilie Heptrevirinae (ehemals „Supergruppe 5“)
- Gattung Caecilivirus
- Gattung Crahelivirus
- Gattung Fipivirus
- Gattung Gruhelivirus
- Gattung Hepatovirus[21][8]
- Spezies Hepatitis-A-Virus (HAV, offiziell Hepatovirus A, HVA)
- Spezies Phopivirus (offiziellHepatovirus B, HVB)
- Spezies Hedgehog hepatovirus (offiziell Hepatovirus H, HVH)
- Gattung Rohelivirus
- Gattung Tremovirus
- „Supergruppe 6“ (noch ohne ICTV-anerkannten Familienstatus)
- Gattung Harkavirus
- Spezies Harkavirus A
- ohne zugeordnete Familie oder Supergruppe
- Gattung Ampivirus
- Spezies Ampivirus A
- Gattung Chipolycivirus
- Spezies Chironomus riparius virus 1
- Spezies Hubei chipolycivirus
- Gattung Hupolycivirus
- Spezies Hubei hupolycivirus
- Gattung Sopolycivirus
- Spezies Formica exsecta virus 3
- Spezies Lasius neglectus virus 1
- Spezies Lasius neglectus virus 2
- Spezies Lasius niger virus 1
- Spezies Linepithema humile virus 2
- Spezies Monomorium pharaonis virus 1
- Spezies Monomorium pharaonis virus 2
- Spezies Myrmica scabrinodis virus 1
- Spezies Shuangao insect virus 8
- Spezies Solenopsis invicta virus 2
- Spezies Solenopsis invicta virus 4
- Spezies Säure-stabiles Equines Picornavirus (Acid-stable Equine Picornavirus, EqPV)[23][25]
- Spezies Aviäres Entero-ähnliches Virus 2 (Avian entero-like virus 2, AELV-2)[26][24]
- Spezies „Aviäres Entero-ähnliches Virus 3“ (AELV-3)[24]
- Spezies „Aviäres Entero-ähnliches Virus 4“ (AELV-4)[24]
- Spezies „Aviäres Nephritis-Virus 3“ (Avians nephritis virus 3, ANV-3)[27][24]
- Spezies „Barramundi-Virus“ („Barramundi virus“, BaV-1)[22][24] (Barramundi, Lates calcarifer)
- Spezies „Entero-ähnliches Virus des Kakadu“ („Cockatoo entero-like virus“, CELV)[26][24]
- Spezies „Gänse-Picornavirus 1“ („Goose picornavirus 1“, GPV-1)
- Spezies „Übertragbares Perlhuhn-Enteritis-Virus“ („Guineafowl transmissible enteritis virus“, GTEV)[26][24]
- Spezies „Picorna-ähnliches Virus des Seehundes“ („Seal picorna-like virus“, SPLV)[23][24] – vgl. auch Anthony et al. (2015)[28] und Krumbholz et al. (2017)[29]
- Spezies „Wolfsbarsch-Virus 1“ („Sea-bass virus“, SBV-1)[24] – Acronym SBV nicht eindeutig, auch für Schmallenberg-Virus))[22]
- Spezies „Sikhote-Alyn-Virus“ („Sikhote-Alyn virus“, SAV)[22][24]
- Spezies „Stint-Virus 1“ („Smelt virus 1“, SmV-1)[22][24] (Stinte, Gattung Osmerus mit Stint)
- Spezies „Stint-Virus 2“ („Smelt virus 2“, SmV-2)[22][24]
- Spezies „Syr-Daria-Valley-Fieber-Virus“ („Syr-Darya Valley fever virus“, SDFV)[22][24]
- Spezies „Steinbutt-Virus 1“ („Turbot virus 1“, TuV-1)[22][24]
- Spezies „Entero-ähnliches Virus der Pute“ („Turkey entero-like virus“, TELV)[26][22][24]
- Spezies „Pseudoenterovirus der Pute 1“ („Turkey pseudo enterovirus 1“, TPEV-1)[26][22][24]
- Spezies „Pseudoenterovirus der Pute 2“ („Turkey pseudo enterovirus 1“, TPEV-2)[26][22][24]
Die Familie Picornaviridae teilt sehr viele Eigenschaften wie beispielsweise die Kapsidarchitektur, die Genomorganisation und phylogenetisch sehr ähnliche virale Proteine mit anderen Virusfamilien. Die Gesamtheit dieser ähnlichen Virusgruppen hat man als Picornavirus-Supergruppe bezeichnet. Aus dieser Gruppe ist inzwischen die Virusordnung Picornavirales der Picornaviridae hervorgegangen.[30][1]
Weblinks
- Picornaviridae: Genera und Spezies (NCBI)
- Bildergalerie der Familie Picornaviridae (Big Picture Book of Viruses)
- Janelle R. Wierenga, Kerri J. Morgan, Harry S. Taylor, Stuart Hunter, Lisa S. Argilla, Trudi Webster, Lauren Lim, Rebecca M. Grimwood, Hendrik Schultz, Fátima Jorge, Mihnea Bostina, Laura Burga, Puawai Swindells-Wallace, Edward C. Holmes, Kate McInnes, Jemma L. Geoghegan: Total infectome investigation of diphtheritic stomatitis in yellow-eyed penguins (Megadyptes antipodes) reveals a novel and abundant megrivirus. Auf: bioRxiv vom 9. Juli 2023; doi:10.1101/2023.07.09.548243 (Preprint, englisch). Das vorgeschlagene „Yellow-eyed penguin megrivirus“ (YEP megrivirus) verursacht augenscheinlich eine der beiden Viruserkrankungen, die das Sterben der Gelbaugenpinguine in Neuseeland (2023) verursachen; evtl. kommt noch ein weiteres „Yellow-eyed penguin picornavirus“ (YEP picornavirus), nahe verwandt mit dem „Gänse-Picornavirus 1“ hinzu. Dazu:
- New Zealand probes mystery illness killing rare penguins. Auf: france24.com vom 22. September 2023. Quelle: AFP (englisch).
- Tiere in Neuseeland unter Druck: Forscher finden wohl Ursache für Pinguin-Sterben. Auf: n-tv.de Wissen vom 22. September 2023.
Literatur
- G. Stanway, F. Brown et al.: Picornaviridae. In: C.M. Fauquet, M.A. Mayo et al.: Eighth Report of the International Committee on Taxonomy of Viruses, London, San Diego, 2005, S. 757–778
- David M. Knipe, Peter M. Howley et al. (Hrsg.): Fields' Virology, 4. Auflage, Philadelphia 2001
- S. Mordow, D. Falke: Molekulare Virologie, Spektrum Akad. Verlag, Heidelberg, Berlin 1997
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Albert Heim: Picornaviren. In: Sebastian Suerbaum, Gerd-Dieter Burchard, Stefan H. E. Kaufmann, Thomas F. Schulz (Hrsg.): Medizinische Mikrobiologie und Infektiologie. Springer-Verlag, 2016, ISBN 978-3-662-48678-8, S. 457–458, doi:10.1007/978-3-662-48678-8_55.
- ViralZone: ICTV 2016 Master Species List #31 with Acronyms, (Excel XLSX), SIB Swiss Institute of Bioinformatics
- Genus supergroups, The Picornavirus Pages (2006–2019), The Pirbright Institute, UK
- ICTV: ICTV Master Species List 2021.v1, New MSL including all taxa updates since the 2020 release, March 2022 (MSL #37)
- SIB: Cardiovirus, auf: ViralZone
- NCBI: Cardiovirus (genus)
- Eda Altan, Kristen Aiemjoy, Tung G. Phan, Xutao Deng, Solomon Aragie, Zerihun Tadesse, Kelly E. Callahan, Jeremy Keenan, Eric Delwart: Enteric virome of Ethiopian children participating in a clean water intervention trial, in: Plos One, 16. August 2018, doi:10.1371/journal.pone.0202054
- NCBI: Gallivirus (genus)
- William Marciel de Souza, Marcılio Jorge Fumagalli, Matheus Cavalheiro Martin, Jansen de Araujo, Maria Angela Orsi, Luiz Francisco Sanfilippo, Sejal Modha, Edison Luiz Durigon, Jose Luiz Proenca-Modena, Clarice Weis Arns, Pablo Ramiro Murcia, Luiz Tadeu Moraes Figueiredo: Pingu virus: A new picornavirus in penguins from Antarctica], in: Virus Evolution 5(2), 2019, doi:10.1093/ve/vez047
- NCBI: Kobuvirus (genus)
- NCBI: Megrivirus (genus)
- Claude Kwe Yinda, Jan Esefeld, Hans-Ulrich Peter, Jelle Matthijnssens, Roland Zell: Penguin megrivirus, a novel picornavirus from an Adélie penguin (Pygoscelis adeliae). In: Archives of Virology, Band 164, Nr. 11, November 2019, S. 2887-2890; doi:10.1007/s00705-019-04404-9, PMID 31494778, ResearchGate, Epub 7. September 2019 (englisch).
- NCBI: Oscivirus (genus)
- NCBI: Passerivirus (genus)
- NCBI: Rafivirus (genus)
- NCBI: Rosavirus (genus)
- NCBI: Sakobuvirus (genus)
- NCBI: Salivirus (genus)
- NCBI: Sicinivirus (genus)
- SIB: Hepatovirus, auf: ViralZone
- ICTV: Family - Picornaviridae (2012) und Picornaviridae (2011), Virus Taxonomy: Ninth Report of the International Committee on Taxonomy of Viruses
- Nick J. Knowles: A Pan-Picornavirus RT-PCR: Identification of Novel Picornavirus Species, Institute for Animal Health (IAH), Pirbright Laboratory, Pirbright, Woking, Surrey, UK (undatiert)
- ABAS: TRBA 462 „Einstufung von Viren in Risikogruppen“, Technische Regeln für Biologische Arbeitsstoffe, Nr. 462, GMBl Nr. 15–20 vom 25. April 2012, letzte Änderung: 3. Juli 2018
- Thomas J. Divers et al.: New Parvovirus Associated with Serum Hepatitis in Horses after Inoculation of Common Biological Product. In: Emerging infectious diseases. Band 24, Nummer 2, 02 2018, S. 303–310, doi:10.3201/eid2402.171031, PMID 29350162, PMC 5782890 (freier Volltext).
- Avian PLV, auf: The Picornavirus Pages
- Sjaak de Wit, Carla Schrier, Gerdy Ten Dam, Yvonne Biermann, Ineke Verstegen, Frans Edens: Detection and characterisation of a new astrovirus in chicken and turkeys with enteric and locomotion disorders, in: Avian Pathology, Taylor & Francis, 2011, S. 1ff, doi:10.1080/03079457.2011.596813, hal-00720583 (Preprint)
- S. J. Anthony, J. A. St. Leger, E. Liang, A. L. Hicks, M. D. Sanchez-Leon, K. Jain, J. H. Lefkowitch, I. Navarrete-Macias, N. Knowles, T. Goldstein, K. Pugliares, H. S. Ip, T. Rowles, and W. I. Lipkina: Discovery of a Novel Hepatovirus (Phopivirus of Seals) Related to Human Hepatitis A Virus. In: mBio. Band 6, Nummer 4, August 2015, S. e01180-15, doi:10.1128/mBio.01180-15, PMID 26307166, PMC 4550696 (freier Volltext).
- Andi Krumbholz, Marco Groth, Jan Esefeld, Hans-Ulrich Peter, Roland Zell: Genome Sequence of a Novel Picorna-Like RNA Virus from Feces of the Antarctic Fur Seal (). In: Genome announcements. Band 5, Nummer 36, September 2017, S. , doi:10.1128/genomeA.01001-17, PMID 28883153, PMC 5589547 (freier Volltext).
- Eugene V. Koonin, Valerian V. Dolja, Mart Krupovic: Origins and evolution of viruses of eukaryotes: The ultimate modularity, in: Virology, Mai 2015; 479-480. 2–25, PMID 25771806, PMC 5898234 (freier Volltext).