Phosphoglucomutase
Phosphoglucomutasen (PGM) sind Enzyme, die die Verschiebung des Phosphatrests in Glucosephosphat von 1- auf 6-Position und umgekehrt katalysieren. Es sind zwei Klassen bekannt: Wenn das Enzym spezifisch für das α-Anomer der Glucosephosphate ist (vgl. unten Reaktionen), nennt man sie α-Phosphoglucomutasen (αPGM). Dagegen akzeptieren β-Phosphoglucomutasen nur β-Anomere (βPGM, EC 5.4.2.6).[1]
| Phosphoglucomutase | ||
|---|---|---|
| Masse/Länge Primärstruktur | 561 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Monomer | |
| Kofaktor | Magnesium | |
| Isoformen | 2 | |
| Bezeichner | ||
| Gen-Name(n) | PGM1, PGM2, PGM3 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 5.4.2.2, Isomerase | |
| Reaktionsart | Umlagerung | |
| Substrat | α-D-Glucose-1-phosphat | |
| Produkte | α-D-Glucose-6-phosphat | |
| Vorkommen | ||
| Homologie-Familie | Hovergen | |
| Übergeordnetes Taxon | Lebewesen | |
Vorkommen
α-Phosphoglucomutasen sind in Prokaryoten und Eukaryoten verbreitet, β-Phosphoglucomutase nur in bestimmten Bakterien und Protisten.
Bedeutung
Diese Umlagerungen sind Teil mehrerer Stoffwechselwege in allen Lebewesen. Im Menschen sind mehrere Gene bekannt, die für verschiedene Homologe codieren, von denen aber nur drei enzymatische Aktivitäten aufweisen: PGM1, PGM2 und PGM3. Letztere ist aber eine Acetylglucosamin-Phosphomutase (EC 5.4.2.3). Mutationen am PGM1- oder PGM2-Gen können zu PGM-Mangel führen. Es sind viele genetische Varianten der PGM bekannt.[2]
Das PGM1-Gen enthält mehrere Hotspots der Rekombination, woraus sich die Vielzahl an Varianten erklärt. Es gibt Hinweise darauf, dass Fehlgeburten auf manche dieser Varianten zurückzuführen sind. Die Aktivität der PGM1 wird durch Bindung an und Phosphorylierung durch Pak1 erhöht.[3][4][5]
Katalysiertes Gleichgewicht
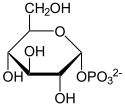

α-Glucose-1-phosphat wird zu Glucose-6-phosphat umgelagert und umgekehrt. Die Reaktionen sind Teil des Glykogen- und Stärkeabbaus, der Glycogensynthese, des Uronsäuren-Stoffwechsels und des Galactose-Stoffwechsels. Bei der Reaktion[6] wird zunächst ein Phosphatrest von einem Phosphoserin im aktiven Zentrum auf die nicht phosphorylierte Stelle übertragen, sodass α-Glucose-1,6-bisphosphat entsteht. Letzteres dreht sich, sodass der andere Phosphatrest vom Serin übernommen werden kann. Das Glucose-1,6-bisphosphat ist relativ fest an das Enzym gebunden, kann sich aber auch lösen, dann muss das Enzym wieder mit Glucose-1,6-bisphosphat aktiviert werden. Eine bakterielle Form der Phosphoglucomutase, EC 5.4.2.5, nutzt Glucose anstelle von Glucose-1,6-bisphosphat als Cofaktor.[7]
Weblinks
Einzelnachweise
- Dai, J. et al. (2006): Conformational cycling in beta-phosphoglucomutase catalysis: reorientation of the beta-D-glucose 1,6-(Bis)phosphate intermediate. In: Biochemistry 45(25); 7818–7824; PMID 16784233; doi:10.1021/bi060136v
- UniProt P36871
- Rana NA, Ebenezer ND, Webster AR, et al: Recombination hotspots and block structure of linkage disequilibrium in the human genome exemplified by detailed analysis of PGM1 on 1p31. In: Hum. Mol. Genet. 13. Jahrgang, Nr. 24, Dezember 2004, S. 3089–102, doi:10.1093/hmg/ddh337, PMID 15509594.
- Gloria-Bottini F, Lucarini N, Palmarino R, et al: Phosphoglucomutase genetic polymorphism of newborns. In: Am. J. Hum. Biol. 13. Jahrgang, Nr. 1, 2001, S. 9–14, doi:10.1002/1520-6300(200101/02)13:1<9::AID-AJHB1001>3.0.CO;2-1, PMID 11466970.
- Gururaj A, Barnes CJ, Vadlamudi RK, Kumar R: Regulation of phosphoglucomutase 1 phosphorylation and activity by a signaling kinase. In: Oncogene. 23. Jahrgang, Nr. 49, Oktober 2004, S. 8118–27, doi:10.1038/sj.onc.1207969, PMID 15378030.
- Natércia F. Brás, Pedro A. Fernandes, Maria J. Ramos, Steven D. Schwartz: Mechanistic Insights on Human Phosphoglucomutase Revealed by Transition Path Sampling and Molecular Dynamics Calculations. In: Chemistry - A European Journal. Band 24, Nr. 8, 6. Februar 2018, S. 1978–1987, doi:10.1002/chem.201705090, PMID 29131453, PMC 5801119 (freier Volltext).
- Atsuko Fujimoto, Peter Ingram, Roberts A. Smith: d-Glucose-1-phosphate: d-glucose-6-phosphotransferase. In: Biochimica et Biophysica Acta (BBA) - Nucleic Acids and Protein Synthesis. Band 96, Nr. 1, Januar 1965, S. 91–101, doi:10.1016/0005-2787(65)90613-1.