Nylonase
Nylonase ist der populärwissenschaftliche Name für eine Klasse von drei Enzymen, die in der Lage sind, die Hydrolyse von Oligomeren der 6-Aminohexansäure (Ahx) zu katalysieren. Die Enzyme konnten aus mehreren Bakterienarten isoliert werden. Der wissenschaftlich korrekte Name für diese Enzyme ist 6-Aminohexanoatoligomerhydrolasen. Sie werden zu den Amidasen gezählt, die wiederum eine Untergruppe der Hydrolasen bilden. Die Substrate für diese Enzyme, die Oligomere der 6-Aminohexansäure, gibt es auf der Erde erst seit dem Jahr 1938. Sie entstehen als Nebenprodukte bei der großtechnischen Herstellung von Polyamid 6 (Perlon) und gelangen so in die Umwelt. Nach dem vorherrschenden naturwissenschaftlichen Konsens erwarben die mit diesen Enzymen ausgestatteten Bakterien die Fähigkeit zur Verdauung der Polyamid-6-Nebenprodukte durch evolutionäre Prozesse. Für Evolutionsbiologen ist die Entwicklung der Nylonasen durch Bakterien ein Paradebeispiel der beobachtbaren Evolution.
Polyamid 6
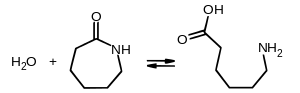
Polyamid 6 (PA6), auch Nylon 6 genannt und besser bekannt unter der Handelsmarke Perlon, wurde erstmals am 29. Januar 1938 von dem deutschen Chemiker Paul Schlack in den Laboren der Forschungsabteilung der Aceta GmbH in Berlin-Lichtenberg hergestellt. Für die Synthese von PA6 nahm Schlack das heute noch dafür verwendete ε-Caprolactam als Ausgangsmaterial. Die Polymerisation geht von 6-Aminohexansäure (Ahx) aus, die aus ε-Caprolactam und Wasser entsteht.
Bildung von 6-Aminohexansäure durch Hydrolyse von ε-Caprolactam
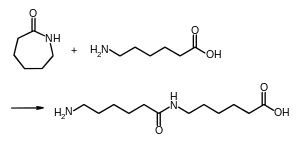
Die 6-Aminohexansäure reagiert dann unter Amidbildung mit einem Molekül ε-Caprolactam zu einem Dimer, dem N-(6-Aminohexanoyl)-6-aminohexanoat, kurz (Ahx2) genannt.
Reaktion von 6-Aminohexansäure mit ε-Caprolactam zu Ahx2
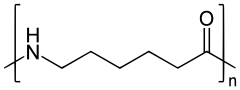
Dieses wiederum kann mit einem weiteren Molekül ε-Caprolactam zu einem Trimer reagieren. Die Polymerisationsreaktion kann so durch Anlagerung weiterer ε-Caprolactam-Moleküle voranschreiten. Polyamid 6 besteht im Wesentlichen aus über 100 miteinander verknüpften Aminohexansäureeinheiten.
Die Struktur von Polyamid 6. Der Buchstabe n steht hierbei für die Anzahl der miteinander verknüpften Einheiten an 6-Aminohexansäure. Der Polymerisationsgrad liegt im Mittel bei n=100.
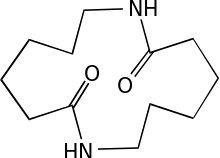
Die Polymerisation ist jedoch ein weitgehend unkontrolliert ablaufender Prozess, der in einer relativ breiten Verteilung der Molmassen resultiert. So finden sich im Polymerisat auch niedermolekulare Verbindungen (Oligomere), wie beispielsweise Dimere (Ahx2), Trimere (Ahx3) usw., der 6-Aminohexansäure. Durch Kopf-Schwanz-Verknüpfung können auch zyklische Oligomere, wie beispielsweise 1,8-Diazacyclotetradecan-2,9-dion entstehen. Diese Oligomere sind unerwünschte Nebenprodukte der Polyamid-6-Produktion.[1]
Die Abwässer der Produktionsbetriebe von Polyamid 6 enthalten neben ε-Caprolactam und 6-Aminohexansäure auch die linearen und zyklischen Oligomere von Polyamid 6. Die Löslichkeit der linearen Oligomere nimmt mit zunehmender Kettenlänge ab. Das Trimer Ahx3 ist zu 1,8 %, das Tetramer Ahx4 zu 0,3 % und das Pentamer Ahx5 nur noch zu 0,01 % in Wasser löslich. Das Hexamer Ahx6 ist in Wasser so gut wie unlöslich.[2][3] Während des Produktionsprozesses werden die Oligomere mit heißem Wasser vom gewünschten Produkt abgetrennt und gelangen so auch ins Abwasser. Beim Abkühlen des Waschwassers fällt ein Großteil der Nebenprodukte als Feststoff aus. Abhängig von der Prozessführung und der Größe der Produktionsanlage fallen so pro Jahr zwischen 20 und 180 Tonnen Oligomere und ε-Caprolactam für einen Betrieb an. Diese Produktionsabfälle werden häufig durch Vergraben in Deponien entsorgt.[4] Weltweit werden pro Jahr etwa 4 Millionen Tonnen Polyamid 6 produziert (Stand 2010).[5]
„Normale“ Mikroorganismen sind nicht in der Lage die Nebenprodukte der Polyamid-6-Produktion abzubauen. Sie können im Wesentlichen nur peptidische Bindungen von α-Aminosäuren mit Hilfe von Peptidasen spalten. Amidbindungen in ε-Position sind erst seit den Versuchen von Schlack, bzw. beim Polyamid 6.6 seit dem 28. Februar 1935 durch Wallace Hume Carothers bekannt.[6]
Nylonasen und die sie produzierenden Bakterien
1965 isolierte der Japaner Takashi Fukumura aus dem Abwasser eines Polyamid-6-produzierendes Betriebes der Firma Toyo Rayon Co., Ltd.[7] (heute Toray) in Nagoya[8] elf verschiedene Bakterienstämme, die in der Lage waren in einem Nährmedium mit 0,6 % ε-Caprolactam zu wachsen. Ein Bakterienstamm (Corynebacterium aurantiacum) konnte darüber hinaus lineare und zyklische Oligomere von 6-Aminohexansäure – mit Ausnahme des zyklischen 6-Aminohexansäure-Dimers[2] – verstoffwechseln (metabolisieren).[9][10][11] Vier Jahre nach Fukumura isolierte eine andere japanische Arbeitsgruppe aus dem gleichen Umfeld ein Bakterium aus der Gattung der Pseudomonaden, das in der Lage ist auch zyklische Oligomere von 6-Aminohexansäure zu metabolisieren.[12][2] Eine Arbeitsgruppe um Hirosuke Okada isolierte 1974 aus Klärschlamm den Bakterienstamm KI72 von Flavobacterium sp.,[# 1][13] der in der Lage ist, mit dem zyklischen Dimer von 6-Aminohexansäure als einziger Kohlenstoff- und Stickstoffquelle zu gedeihen.[2][14] Der zwei Jahre zuvor von derselben Arbeitsgruppe isolierte Stamm KF71 des Flavobakteriums konnte dies nicht.[15] KI72 ist in der Lage, ε-Caprolactam, 6-Aminohexansäure und das zyklische Ahx-Dimer sowie die linearen Di- bis Hexamere von 6-Aminohexansäure zu metabolisieren. Aus dem Bakterium isolierten die Forscher zwei Enzyme, von denen eines die Hydrolyse des zyklischen 6-Aminohexansäure-Dimers zu Ahx2 katalysiert. Das andere Enzym katalysiert die Hydrolyse der linearen Oligomere.[2] Das Enzym, das das zyklische 6-Aminohexansäure-Dimer spalten kann, wurde auf seine hydrolytischen Fähigkeiten gegenüber anderen, natürlichen Substraten getestet. Überraschenderweise konnte es bei keinem der 50 Dipeptide und 16 Tripeptide die Hydrolyse katalysieren.[14] In nachfolgenden Versuchen erhöhte sich die Zahl der natürlichen Verbindungen mit Amidbindung, die kein Substrat für dieses NylA genannte Enzym bilden, auf über 100.[16] Auch für das Enzym, das als Substrat die linearen Oligomere hat und heute NylB genannt wird, konnte kein „natürliches“ Substrat gefunden werden.[17][16]
Das Zelllysat, das von Zellen erhalten wurde deren Nährmedium kein zyklisches Dimer enthielt, war nicht in der Lage die Hydrolyse des zyklischen Dimers zu katalysieren. Im Gegensatz zu dem Zelllysat das von Zellen mit zyklischem Dimer als Nährmedium erhalten wurde. Okada und seine Mitautoren schlossen aus diesem Verhalten, dass die 6-Aminohexansäure-Dimer-Hydrolase, ein induzierbares Enzym ist, das heißt, dass die Konzentration des Enzyms durch die Anwesenheit eines Induktors – in diesem Fall des zyklischen Dimers – erhöht wird.[14]
Es gibt zwei mögliche Ursachen dafür, warum ein Enzym eine Aktivität gegenüber einem unnatürlichen Substrat aufweist. Im ersten, einfacheren Fall ist das unnatürliche Substrat ein Analogon zum natürlichen Substrat. Im zweiten Fall wird die Katalyse durch ein evolutionär entwickeltes Enzym bewerkstelligt, das seine ursprüngliche Aktivität gegenüber einem natürlichen Substrat durch Mutation verloren hat.[14] Da Okada und seine Mitautoren in ihren Versuchen kein natürliches Substrat für die 6-Aminohexansäure-Dimer-Hydrolase fanden und dieses Enzym im Vergleich zu anderen zyklischen Amidhydrolasen, wie beispielsweise Penicillinase (>35 s−1), eine vergleichsweise niedrige katalytische Konstante kcat (8 s−1) aufweist, schlossen sie, dass die 6-Aminohexansäure-Dimer-Hydrolase wahrscheinlich durch evolutionäre Anpassung entstanden ist.[14]
1992 isolierte Seiji Negoro mit weiteren Kollegen aus Okadas Arbeitsgruppe ein drittes Enzym, heute NylC genannt, aus dem Bakterienstamm.[18] NylC ist eine Endohydrolase, das bedeutet, dass das Enzym die Hydrolyse im Inneren (griechisch ἔνδον endon ‚innen‘) der Oligomerkette katalysiert. Gegenüber dem zyklischen Dimer hat es keine Aktivität, aber gegenüber dem zyklischen Tetramer und Pentamer.[16] Auch bei NylC konnte keine Aktivität gegenüber „natürlichen“ Amidbindungen festgestellt werden.[18]
Für den Abbau der 6-Aminohexansäure-Oligomere sind somit insgesamt drei Enzyme verantwortlich:[19][20][16]
- zyklische 6-Aminohexansäure-Dimer-Hydrolase (NylA)[14] EC 3.5.2.12
- 6-Aminohexansäure-Dimer-Hydrolase (NylB)[17] EC 3.5.1.46
- 6-Aminohexanoat-Oligomer-Endohydrolase (NylC)[18] EC 3.5.1.117

Vergleich der Eigenschaften der drei Polyamid-6-abbauenden Enzyme von Flavobacterium sp. KI72:[16]
| Eigenschaften | Enzym | ||
|---|---|---|---|
| Gen | nylA | nylB | nylC |
| Molare Masse | 52 kDa | 42 kDa | 37 kDa |
| Anzahl an Aminosäuren | 493 | 392 | 355 |
| optimaler pH-Wert | 7,4 | 9,0 | 7,0 |
| Temperaturoptimum | 34 °C | 40 °C | 42 °C |
| Spezifische Aktivität gegenüber | |||
| Ahx2 (lineares Dimer) | <0,001 | 0,94 | 0,00044 |
| Ahx3 (lineares Trimer) | <0,001 | 0,75 | 0,11 |
| Ahx4 (lineares Tetramer) | <0,001 | 0,57 | 0,42 |
| Ahx5 (lineares Pentamer) | <0,001 | 0,24 | 0,47 |
| cyclo(Ahx)2 (zyklisches Dimer) | 2,8 | <0,0001 | <0,0001 |
| cyclo(Ahx)4 (zyklisches Tetramer) | <0,001 | <0,0001 | 0,36 |
| ε-Caprolactam | <0,001 | <0,0001 | <0,0001 |
Die Aktivitätswerte wurden bei einer Substratkonzentration von 1 mM erhalten und sind in mol NH2 pro min und mg aufgereinigtem Protein (U/mg) angegeben. Bei den grün hinterlegten Werten ist eine signifikante Aktivität gegenüber dem jeweiligen Substrat gegeben. [16]
NylA
Die zyklische 6-Aminohexansäure-Dimer-Hydrolase (NylA) katalysiert spezifisch die Hydrolyse einer der beiden äquivalenten Amidbindungen in 1,8-Diazacyclotetradecan-2,9-dion, dem zyklischen Dimer der 6-Aminohexansäure. Dabei entsteht N-(6-Aminohexanoyl)-6-aminohexanoat, das lineare Dimer der 6-Aminohexansäure. NylA gehört zur Amidase-Signatur-Familie (engl. amidase signature family).[23][24] Die Mitglieder dieser über 200 Enzyme umfassenden Familie haben einen konservierten Sequenzbereich von 160 Aminosäuren, die Amidase-Signatur.[25] Gegenüber anderen Polyamid-6-Oligomeren weist NylA so gut wie keine Aktivität auf.
NylB
Die 6-Aminohexansäure-Dimer-Hydrolase (NylB) hat in ihrem aktiven Zentrum die katalytische Triade Ser112/Lys115/Tyr215 und ein Oxyanion-Loch. Die katalytische Triade entspricht der einiger anderer bakterieller Serinhydrolasen, wie beispielsweise D-Alanin-Transpeptidase,[27] Carboxylesterase 2 (estB) und Klasse C Serin-β-Lactamasen.[28] Ein wesentlicher Unterschied ist jedoch die Aminosäure Tyrosin in Position 170 (Tyr170), die sich so bei keiner Serin-β-Lactamasen findet.[29] Tyr170 hat durch seine Interaktion mit der katalytischen Triade einen wesentlichen Anteil an der Fähigkeit von NylB die Hydrolyse von Ahx2 zu katalysieren. Die Substratbindung im Enzym wird durch Tyr170 erhöht.[30][31] Außerdem fördert Tyr170 im Enzym – durch Störung eines tetraedrischen Übergangszustand – eine Konformationsänderung des Substrates, die günstig für dessen Protonierung durch Tyr215 von der katalytischen Triade ist. Wird das Tyrosin in Position 170 durch das sterisch sehr ähnliche Phenylalanin ersetzt (Tyr170Phe), so sinkt die Aktivität von NylB gegenüber Ahx2 auf 1,4 %.[21][31]
Die Aktivität von NylB gegenüber linearen Polyamid-6-Oligomeren nimmt mit zunehmender Kettenlänge deutlich ab. Beim Hexamer (Ahx6) beträgt die Aktivität nur noch 8 % des Wertes vom Dimer (Ahx2), beim Eikosamer (Ahx20) sinkt der Wert auf 0,3 %.[32]
Wie andere Serinproteasen zeigt NylB auch Eigenschaften einer Esterase und ist in der Lage die Hydrolyse verschiedener Carbonsäureester zu katalysieren.[19][33]
NylB′
Von NylB gibt es ein Homolog, das NylB′ genannt wird. Beide Enzyme bestehen aus 392 Aminosäuren, die sich aber an 46 Positionen unterscheiden. NylB′ hat nur etwa 0,5 % der Aktivität von NylB. 2009 konnte eine japanische Arbeitsgruppe mittels molekularem Design zeigen, dass durch den gezielten Austausch von drei Aminosäuren die katalytischen Eigenschaften NylB′ um den Faktor 160 gesteigert werden können.[34]
NylC
Die 6-Aminohexanoat-Oligomer-Endohydrolase (NylC) katalysiert die Hydrolyse von linearen und zyklischen Polyamid-6-Oligomeren mit mehr als drei 6-Aminohexansäure-Einheiten. Die Aktivität gegenüber dem linearen und zyklischen 6-Aminohexansäure-Dimer ist sehr gering. Das Enzym wurde unter anderem aus Kulturen von Arthrobacter (NylCp2), Agromyces (NylCA) und Kocuria (NylCK) isoliert. NylCA und NylCK unterscheiden sich durch 5 bis 15 Aminosäuresubstitutionen von NylCp2 und sind um 10 bis 20 K thermostabiler. In wässriger Lösung bildet NylC Oligomere mit einer mittleren molaren Masse von ca. 93 kDa. Auch Röntgenstrukturanalysen sprechen für eine dimere bzw. trimere Struktur.[23]
Evolution im Labor

Der Bakterienstamm Pseudomonas aeruginosa PAO, der ursprünglich in Neuseeland isoliert wurde, ist biochemisch und genetisch gut erforscht und der Standardstamm dieser Bakterienart. Pseudomonas aeruginosa PAO zeigt keinerlei Aktivität gegenüber dem linearen Dimer Ahx2, sowie anderen Nebenprodukten der Polyamid-6-Produktion.[36] 1995 kultivierte eine Arbeitsgruppe an der Universität Ōsaka Pseudomonas aeruginosa PAO in dem Minimalmedium M9, das im Versuch aus 2 g/l Glucose und 1 g/l Ammoniumchlorid bestand. Verschiedene Verdünnungen dieser Kolonie gaben sie auf Kulturschalen, die Ahx2 in einer Konzentration von 2 g/l als einzige Kohlenstoff- und Stickstoffquelle enthielten. Die Polyamid-6-Oligomere sind für diese Bakterienart untoxisch. Nach neun Tagen Inkubation bei 30 °C erhielten die Experimentatoren mit einer Häufigkeit von 0,1 % Kolonien mit einem ausgeprägten Wachstum („hypergrowing colonies“). Die Bakterien dieser Kolonien hatten in dieser Zeit die Fähigkeit erworben das lineare Dimer der 6-Aminohexansäure als Nährstoff zu nutzen.[37] Eine dieser Kolonien, die PAO5502 genannt wurde, war nach drei Monaten Inkubation in der Lage auch das zyklische Dimer der 6-Aminohexansäure zu metabolisieren. Die Wachstumsrate lag bei 0,1 h−1 im linearen und bei 0,03 h−1 im zyklischen Dimer. PAO5502 hydrolysiert im ersten Schritt das zyklische zum linearen Dimer und im zweiten Schritt das lineare Dimer in zwei Moleküle 6-Aminohexansäure.[36]
Durch Nährstoffmangel kann in Bakterien die Mutationsrate um Faktoren im Bereich von 10 000 erhöht werden.[38] Da diese Bedingungen bei der Inkubation von Pseudomonas aeruginosa vorlagen, ist davon auszugehen, dass dies ein entscheidender Faktor für den schnellen Erwerb der neuen Fähigkeiten von Pseudomonas aeruginosa war.[36]
ε-Caprolactam als Substrat
1998 wurde mit Pseudomonas aeruginosa MCM B-407 aus dem Belebtschlamm einer Kläranlage eines Polyamid-6-produzierenden Unternehmens ein Bakterienstamm isoliert, der in der Lage ist ε-Caprolactam abzubauen.[39] 2002 wurde diese Eigenschaft auch bei Stämmen von Alcaligenes faecalis, Arthrobacter citrus, Bacillus sphaericus und Rhodococcus rhodochrous nachgewiesen. Der Stamm Alcaligenes faecalis G wächst bei Konzentrationen bis zu 15 g ε-Caprolactam pro Liter und baut es zu 95 bis 97 % innerhalb von 24 bis 48 Stunden ab. Für den effizienten Abbau benötigt es als Nährsalze Magnesium-, Kalium- und Phosphat-Ionen.[40] Bei Proteus sp. und Bordetella sp., die aus Abfalldeponien im Bundesstaat Lagos (Nigeria) isoliert wurden, konnten 2013 ähnliche Abbauraten gemessen werden.[41] Die großen Mengen ε-Caprolactam im Abwasser der Polyamd-6-Produktionsbetriebe belasten die Umwelt beträchtlich. ε-Caprolactam-abbauende Bakterienstämme haben daher eine besondere Bedeutung in der Behandlung solcher Abwässer.[42]
Herkunft der Enzyme

Mit der Entdeckung der Nylonasen stellte sich unmittelbar die Frage, woher diese Enzyme stammen. Schließlich gab es die von dem Flavobacterium sp. KI72 verwerteten Oligomere der 6-Aminohexansäure erst seit etwa 30 Jahren und andere, natürliche Substrate für diese Enzyme wurden nicht gefunden.
Flavobacterium sp. KI72 hat drei Arten von Plasmiden: pOPAD1, pOAD2 und pOAD3.[16] Plasmide sind kleine, meist ringförmige, doppelsträngige DNA-Moleküle, die nicht zum Bakterienchromosom zählen. Produziert werden die drei Polyamid-6-abbauenden Enzyme im Plasmid pOAD2. Dieses Plasmid besteht aus 45.519 Basenpaaren, von denen Guanin und Cytosin etwa zwei Drittel stellen. Das Plasmid enthält die vier Gene nylA, nylB, nylC und nylB′, sowie fünfmal die Insertionssequenz IS6100. Die Sequenz von pOAD2 weist eine signifikante Ähnlichkeit mit der der Gene von penDE (Isopenicillin-N-Acyltransferase), rep (Plasmidinkompatibilität), ftsX (filamentation temperature sensitive) und oppA-F (Oligopeptid-transportierende ATPase) auf.[43]
Susumu Ohno postulierte 1984, dass die Nylonase B durch Genduplikation und nachfolgenden Frameshift entstanden sei.[37] Die Quelle für das einzigartige Protein sei ein ungenutzter offener Leserahmen der vorhandenen kodierenden Sequenz. Nur Sequenzen die mit mehreren Wiederholungen beginnen, seien wahrscheinlich in der Lage abweichende, lange offene Leserahmen bestehen zu lassen. Aus der Basensequenz von NylB im pOAD2-Plasmid von Flavobacterium sp. K172 schloss er, dass das 392 Aminosäuren umfassende Genprodukt (NylB) von einem anderen, vorher existierenden, offenen Leserahmen stammt, der ursprünglich ein 472 Aminosäuren umfassendes Arginin-reiches Protein kodierte.[44] Auch der US-amerikanische Kenneth Miller geht in seinem im Jahr 2008 erschienenen Buch Only a Theory davon aus, dass ein Frameshift in einem Gen der Entstehungsort für die Nylonasen war. Über diese Mutation und nachfolgende Selektion über den Selektionsvorteil Verwertung von Polyamid-6-Oligomeren seien die Bakterienstämme entstanden, die in der Lage sind Polyamid-6-Oligomere abzubauen. Da die Aktivitäten der Nylonasen vergleichsweise gering sind, geht er davon aus, dass der Evolutionsprozess dieser Enzyme noch in vollem Gang ist.[37] Frameshifts sind eine radikale und bei Bakterien vergleichsweise häufig anzutreffende Quelle für Gene mit neuen Eigenschaften.[45] Selbst im menschlichen Genom wurden 470 Frameshiftereignisse nachgewiesen,[46] die zu Proteinen mit neuen Eigenschaften führten.[47]
Zu einem anderen Ergebnis als die Spekulationen um einen Frameshift führen die Ergebnisse einer Arbeitsgruppe um Seiji Negoro von der Präfekturuniversität Hyōgo. Durch Röntgenstrukturanalyse von NylB kommt sie zu dem Resultat, dass dieses Enzym seinen Ursprung in einer Esterase mit β-Lactamase-Faltung hat.[21]
Nylonase und Kreationismus
Nach Auffassung von Kreationisten, wie beispielsweise John N. Moore von der Creation Research Society, sind Genmutationen nicht in der Lage neue Eigenschaften bei einem Organismus zu bewirken. Nach ihrer Meinung bewirken Genmutationen lediglich Veränderungen von bereits existierenden oder bekannten Eigenschaften.[# 2] Auch nach William Dembski, einem Vertreter des Intelligent Designs, ist die Komplexität von Proteinen zu hoch, als dass durch Mutation und Selektion neue Informationen in das Genom aufgenommen werden können. Mutationen würden somit nur Varietäten, nicht aber neue Fähigkeiten hervorrufen können.[48] Neue Eigenschaften eines Organismus lassen sich im Weltbild der Intelligent-Design-Vertreter nur durch einen intelligenten Designer („Gott“) erklären und nicht durch die dazu im Widerspruch stehende Evolution, mit ihren Werkzeugen Mutation und Selektion.
Dies steht in krassem Widerspruch zur Auffassung der meisten Evolutionsbiologen, nach der Mutationen zu neuen, vorteilhaften Eigenschaften führen können und diese Eigenschaften an nachfolgende Generationen vererbt werden können.[49] Die durch Mutationen erworbene Fähigkeit verschiedener Bakterienarten „unnatürliche“ Substanzen wie die Polyamid-6-Oligomere als Nährstoff nutzen zu können, ist für sie – ähnlich wie das E. coli-Langzeitexperiment von Richard Lenski – ein Paradebeispiel der beobachtbaren Evolution.[50] In der Folge entstand um die Nylonasen eine Kontroverse zwischen Evolutionsbiologen und Vertretern des Intelligent Design. Nach Meinung der Vertreter des Intelligent Design ist die Fähigkeit zur Metabolisierung von Polyamid-6-Oligomeren „keine neue Information“ (“Nylonase isn’t new information”).[51]
Weblinks
- William M. Thwaites: New Proteins Without God's Help. In: Creation Evolution Journal. Band 4, Nummer 2, 1985, S. 1–3. (englisch)
- Evolution and Information: The Nylon Bug New Mexicans for Science and Reason (englisch)
- Ker Than: Why scientists dismiss 'intelligent design'. In: nbcnews.com. 23. September 2005, abgerufen am 30. September 2015 (englisch).
Anmerkungen
- Das Bakterium wurde ursprünglich von den Autoren als Achromobacter guttatus klassifiziert.
- Zitat: “Any gene mutation results in no more than alteration of already existing or known traits.” bzw. “Mutations are sources only of differences of characteristic expressions of traits already in existence, and not a source of new traits. Mutations result only in changes within the existing genetic structure.” John N. Moore, Roger J. Cuffey: Paleontologic evidence and organic evolution. In: Journal of the American Scientific Affiliation. Band 24, 1972, S. 160–176.
Einzelnachweise
- Seiji Negoro: Biodegradation of Nylon and other Synthetic Polyamides. (Memento des vom 26. September 2015 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. In: S. Matsumura, A. Steinbüchel (Hrsg.): Miscellaneous biopolymers and biodegradation of synthetic polymers. Band 9, 2005, doi:10.1002/3527600035.bpol9018, ISBN 3-527-30228-X, S. 395–415.
- Shinichi Kinoshita, Sadao Kageyama, Kazuhiko Iba, Yasuhiro Yamada, Hirosuke Okada: Utilization of a Cyclic Dimer and Linear Oligomers of ε-Aminocaproic Acid by Achromobacter guttatus KI 72. In: Agricultural and Biological Chemistry. Band 39, Nummer 6, 1975, S. 1219–1223, doi:10.1271/bbb1961.39.1219
- E. Tsuchida, I. Shinohara, Oligomer and Oligomerization. In: Journal of Synthetic Organic Chemistry Japan. Band 22, Nummer 33, 1964, doi:10.5059/yukigoseikyokaishi.22.33 (Artikel in japanisch)
- N. N. Baxi, A. K. Shah: Biological treatment of the components of solid oligomeric waste from a nylon-6 production plant. In: World Journal of Microbiology and Biotechnology. Band 16, 2000, S. 835–840.
- Polyamide 6. In: pcinylon.com. Archiviert vom (nicht mehr online verfügbar) am 9. Juli 2015; abgerufen am 5. September 2015 (englisch). Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- Joachim Schreiber: Plastisch, Elastisch, und Fantastisch. John Wiley & Sons, 2013, ISBN 978-3-527-66532-7, S. 88 (eingeschränkte Vorschau in der Google-Buchsuche).
- H. Iizuka, T. Tanabe, T. Fukumura, K. Kato: Taxonomic study on the ε-caprolactam-utilizing bacteria. In: The Journal of General and Applied Microbiology. 13, 1967, S. 125–137, doi:10.2323/jgam.13.125.
- Rhodococcus sp. JCM Catalogue
- T. Fukumura: Bacterial breakdown of ε-caprolactam and its cyclic oligomers. In: Plant and Cell Physiology. Band 7, 1966, S. 93–104. (Abstract)
- T. Fukumura: Two bacterial enzymes hydrolyzing oligomers of 6-aminocaproic acid. In: Journal of Biochemistry. Band 59, Nummer 6, 1966, S. 537–544.
- T. Fukumura: Splitting of ε-caprolactam and other lactams by bacteria. In: Plant and Cell Physiology. Band 7, Nummer 1, 1966, S. 105–114. (Abstract)
- N. Maekawa, N. Takenaka, R. Kihara; In: Abst. Ann. Meet., Soc. Ferment. Technol. 1969, S. 8.
- H. Okada, S. Negoro u. a.: Evolutionary adaptation of plasmid-encoded enzymes for degrading nylon oligomers. In: Nature. Band 306, Nummer 5939, 1983, S. 203–206, PMID 6646204.
- S. Kinoshita, S. Negoro u. a.: 6-Aminohexanoic acid cyclic dimer hydrolase. A new cyclic amide hydrolase produced by Achromobacter guttatus KI74. In: [[FEBS Journal|European Journal of Biochemistry]]. Band 80, Nummer 2, 1977, S. 489–495, PMID 923591.
- S. Kinoshita, E. Kobayashi, H. Okada: Degradation of ε-caprolactam by Achromobacter guttatus KF 71. In: Journal of fermentation technology. Band 51, 1973, S. 719.
- S. Negoro: Biodegradation of nylon oligomers. In: Applied Microbiology and Biotechnology. Band 54, Nummer 4, 2000, S. 461–466, PMID 11092619 (Review).
- S. Kinoshita, T. Terada u. a.: Purification and characterization of 6-aminohexanoic-acid-oligomer hydrolase of Flavobacterium sp. Ki72. In: [[FEBS Journal|European Journal of Biochemistry]]. Band 116, Nummer 3, 1981, S. 547–551, PMID 7262074.
- S. Negoro, S. Kakudo u. a.: A new nylon oligomer degradation gene (nylC) on plasmid pOAD2 from a Flavobacterium sp. In: Journal of bacteriology. Band 174, Nummer 24, 1992, S. 7948–7953, PMID 1459943, PMC 207530 (freier Volltext).
- T. Ohki, Y. Wakitani u. a.: Mutational analysis of 6-aminohexanoate-dimer hydrolase: relationship between nylon oligomer hydrolytic and esterolytic activities. In: FEBS Letters. Band 580, Nummer 21, 2006, S. 5054–5058, doi:10.1016/j.febslet.2006.08.031, PMID 16949580.
- S. Negoro, N. Shibata u. a.: Three-dimensional structure of nylon hydrolase and mechanism of nylon-6 hydrolysis. In: The Journal of biological chemistry. Band 287, Nummer 7, 2012, S. 5079–5090, doi:10.1074/jbc.M111.321992, PMID 22187439, PMC 3281642 (freier Volltext).
- S. Negoro, T. Ohki u. a.: Nylon-oligomer degrading enzyme/substrate complex: catalytic mechanism of 6-aminohexanoate-dimer hydrolase. In: Journal of molecular biology. Band 370, Nummer 1, 2007, S. 142–156, doi:10.1016/j.jmb.2007.04.043, PMID 17512009.
- PDB 3A2P
- K. Yasuhira, N. Shibata u. a.: Crystallization and X-ray diffraction analysis of nylon-oligomer hydrolase (NylC) from Agromyces sp. KY5R. In: Acta crystallographica. Section F, Structural biology and crystallization communications. Band 67, Pt 8August 2011, S. 892–895, doi:10.1107/S1744309111022858, PMID 21821888, PMC 3151121 (freier Volltext).
- K. Yasuhira, Y. Uedo u. a.: Genetic organization of nylon-oligomer-degrading enzymes from alkalophilic bacterium, Agromyces sp. KY5R. In: Journal of bioscience and bioengineering. Band 104, Nummer 6, Dezember 2007, S. 521–524, doi:10.1263/jbb.104.521, PMID 18215642.
- Jörg Labahn: Protonierung und biologische Funktion – Die Grenzen der Röntgenstrukturanalyse. Habilitationsschrift, Heinrich-Heine-Universität Düsseldorf, 2003, S. 6.
- PDB 2ZLY
- N. R. Silvaggi, J. W. Anderson u. a.: The crystal structure of phosphonate-inhibited D-Ala-D-Ala peptidase reveals an analogue of a tetrahedral transition state. In: Biochemistry. Band 42, Nummer 5, Februar 2003, S. 1199–1208, doi:10.1021/bi0268955, PMID 12564922.
- T. Ohki, N. Shibata u. a.: Two alternative modes for optimizing nylon-6 byproduct hydrolytic activity from a carboxylesterase with a beta-lactamase fold: X-ray crystallographic analysis of directly evolved 6-aminohexanoate-dimer hydrolase. In: Protein science : a publication of the Protein Society. Band 18, Nummer 8, 2009, S. 1662–1673, doi:10.1002/pro.185, PMID 19521995, PMC 2776954 (freier Volltext).
- S. Negoro, T. Ohki u. a.: X-ray crystallographic analysis of 6-aminohexanoate-dimer hydrolase: molecular basis for the birth of a nylon oligomer-degrading enzyme. In: The Journal of biological chemistry. Band 280, Nummer 47, 2005, S. 39644–39652, doi:10.1074/jbc.M505946200, PMID 16162506.
- Takeshi Baba, Katsumasa Kamiya u. a.: Molecular dynamics studies on the mutational structures of a nylon-6 byproduct-degrading enzyme. In: Chemical Physics Letters. 507, 2011, S. 157–161, doi:10.1016/j.cplett.2011.03.046.
- K. Kamiya, T. Baba u. a.: Nylon-Oligomer Hydrolase Promoting Cleavage Reactions in Unnatural Amide Compounds. In: The journal of physical chemistry letters. Band 5, Nummer 7, 2014, S. 1210–1216, doi:10.1021/jz500323y, PMID 26274473.
- H. Okada, S. Negoro, S. Kinoshita: Enzymes active on unnatural synthetic compounds: nylon oligomer hydrolases controlled by a plasmid and their cloning. In: Ichiro Chibata: Enzyme Engineering. Springer Science & Business Media, 2013, ISBN 978-1-4615-9290-7, S. 491–500 (eingeschränkte Vorschau in der Google-Buchsuche).
- S. Negoro, T. Mitamura u. a.: Determination of the active-site serine of 6-aminohexanoate-dimer hydrolase. In: European Journal of Biochemistry / FEBS. Band 185, Nummer 3, 1989, S. 521–524, PMID 2512123.
- Y. Kawashima, T. Ohki u. a.: Molecular design of a nylon-6 byproduct-degrading enzyme from a carboxylesterase with a beta-lactamase fold. In: The FEBS journal. Band 276, Nummer 9, Mai 2009, S. 2547–2556, doi:10.1111/j.1742-4658.2009.06978.x, PMID 19476493.
- PDB 3AXG
- I. D. Prijambada, S. Negoro u. a.: Emergence of nylon oligomer degradation enzymes in Pseudomonas aeruginosa PAO through experimental evolution. In: Applied and environmental microbiology. Band 61, Nummer 5, 1995, S. 2020–2022, PMID 7646041, PMC 167468 (freier Volltext).
- Kenneth R. Miller: Only a Theory. Penguin, 2008, ISBN 978-1-4406-3403-1, S. 64–68 (eingeschränkte Vorschau in der Google-Buchsuche).
- R. E. Lenski, J. E. Mittler: The directed mutation controversy and neo-Darwinism. In: Science. Band 259, Nummer 5092, 1993, S. 188–194, PMID 7678468 (Review).
- R. S. Kulkarni, P. P. Kanekar: Bioremediation of epsilon-caprolactam from nylon-6 waste water by use of Pseudomonas aeruginosa MCM B-407. In: Current microbiology. Band 37, Nummer 3, 1998, S. 191–194, PMID 9688819.
- N. N. Baxi, A. K. Shah: ε-Caprolactam-degradation by Alcaligenes faecalis for bioremediation of wastewater of a nylon-6 production plant. In: Biotechnology Letters. Band 24, Nummer 14, 2002, S. 1177–1180, doi:10.1023/A:1016187103682.
- H. A. Sanuth, A. Yadav u. a.: ε-Caprolactam Utilization by Proteus sp. and Bordetella sp. Isolated From Solid Waste Dumpsites in Lagos State, Nigeria, First Report. In: Indian journal of microbiology. Band 53, Nummer 2, 2013, S. 221–226, doi:10.1007/s12088-013-0356-5, PMID 24426112, PMC 3626951 (freier Volltext).
- V. Johnson, S. J. Patel u. a.: Caprolactam waste liquor degradation by various yeasts. In: World journal of microbiology & biotechnology. Band 10, Nummer 5, 1994, S. 524–526, doi:10.1007/BF00367658, PMID 24421125.
- K. Kato, K. Ohtsuki u. a.: A plasmid encoding enzymes for nylon oligomer degradation: nucleotide sequence and analysis of pOAD2. In: Microbiology. Band 141 (Pt 10), 1995, S. 2585–2590, doi:10.1099/13500872-141-10-2585, PMID 7582019.
- S. Ohno: Birth of a unique enzyme from an alternative reading frame of the preexisted, internally repetitious coding sequence. In: PNAS. Band 81, Nummer 8, 1984, S. 2421–2425, PMID 6585807, PMC 345072 (freier Volltext).
- A. L. Koch: Catastrophe and what to do about it if you are a bacterium: the importance of frameshift mutants. In: Critical reviews in microbiology. Band 30, Nummer 1, 2004, S. 1–6, doi:10.1080/10408410490266401, PMID 15116759 (Review).
- K. Okamura, L. Feuk u. a.: Frequent appearance of novel protein-coding sequences by frameshift translation. In: Genomics. Band 88, Nummer 6, Dezember 2006, S. 690–697, doi:10.1016/j.ygeno.2006.06.009, PMID 16890400.
- Manyuan Long: Evolution of new genes. In: Jonathan B. Losos, David A. Baum u. a.: The Princeton Guide to Evolution. Princeton University Press, 2013, ISBN 978-1-4008-4806-5, S. 407 (eingeschränkte Vorschau in der Google-Buchsuche).
- Alvin Allison: From Monkeys to Men and Back. AuthorHouse, 2008, ISBN 978-1-4389-3878-3, S. 110 (eingeschränkte Vorschau in der Google-Buchsuche).
- Mutation. In: Science of Everyday Things. 2002. abgerufen am 15. September 2015
- Mark Isaak: The Counter-creationism Handbook. University of California Press, 2007, ISBN 978-0-520-24926-4, S. 305 (eingeschränkte Vorschau in der Google-Buchsuche).
- Nylonase Isn’t New Information. In: evolutiondismantled.com undatiert, abgerufen am 15. September 2015