Gene Drive
Gene Drive (engl. für Genantrieb) bezeichnet Methoden zur beschleunigten Ausbreitung von Genen in Populationen. Während ein Gen, das nur einmal im Genom vorkommt, normalerweise an 50 % der Nachkommen weitergegeben wird, sind es beim Gene Drive 100 %. Gene-Drive-Technologien wurden erstmals 2003 von Austin Burt vorgeschlagen[1] und bisher vor allem in Hefen, Stechmücken und Fruchtfliegen angewendet.

Prinzip
Gene Drive erzeugt eine überproportionale Vererbung in Lebewesen, die eine Entwicklungsstufe mit haploidem Genom (einfachem Chromosomensatz) durchlaufen. Bei Tieren ist diese Entwicklungsstufe die Meiose, sie besitzen die meiste Zeit jedoch einen doppelten Chromosomensatz. Gene Drive erfolgt mittels eines Enzyms (eine Endonuklease), das eine bestimmte DNA-Sequenz aus dem zweiten Strang eines Chromosoms ohne Gene Drive schneidet. Über eine folgende DNA-Reparatur dient der Strang mit Gene Drive (ein Chromosom hat zwei DNA-Stränge) als Reparaturvorlage für die geschnittenen DNA-Stränge und die Vorlage wird auf den anderen Strang kopiert. Dadurch kommt es zur überproportionalen Ausbreitung, denn alle Nachkommen tragen die per Gene Drive „reparierte“ Gensequenz.
Gene Drive kann unter anderem zum Einfügen neuer Gene verwendet werden. Wenn allerdings durch zwei Schnitte eine DNA-Sequenz entfernt wird, und die entfernte DNA-Sequenz essentiell war, entsteht ein Letalfaktor bei allen Nachkommen. Die Elterngeneration wird unfruchtbar, weil die Nachkommen während der Entwicklung sterben. Gene-Drive-Systeme werden eingesetzt, um einzelne Gene entweder analog zur Gentherapie zur Ergänzung des Genoms oder zur Erzeugung einer Unfruchtbarkeit in eine Population einzuführen.[2]
Mechanismus
Verschiedene Methoden werden verwendet, um die Verbreitung von Genen zu erhöhen, z. B. modifizierte eigennützige DNA wie Homing-Endonukleasen,[1] das MEDEA und eine modifizierte CRISPR/Cas-Methode.[3][4]
Letalfaktor
Zuerst wird ein Genome Editing-Mechanismus (eine sequenzspezifische Endonuklease) erzeugt, der die Ziel-DNA-Sequenz bindet und schneidet,[1] möglichst nur an einer Sequenz im Genom (auf zwei Allelen). Alternativ kann auch eine neue Schnittstelle der Endonuklease in das Genom eingefügt werden, die dort sonst nicht vorkommt. Anschließend wird das Gen für diesen Mechanismus in die Ziel-DNA-Sequenz auf dem ersten von zwei Chromosomen eingefügt. Die Ziel-DNA ist dabei ein essentielles Gen, in das der Genome Editing-Mechanismus eingefügt wurde, wodurch das essentielle Gen geteilt und inaktiviert wird.[1] Bei natürlich vorkommender eigennütziger DNA liegt dagegen ein Gruppe-I-Intron oder ein Intein vor, wodurch das umliegende Gen nicht inaktiviert wird.[1] Gleichzeitig liegt das Gen in einer der Erkennungssequenzen der Endonuklease, wodurch ein Herausschneiden des Gens der Endonuklease verhindert wird.[1] Als Ziel-DNA-Sequenz wird zur Sterilisierung ein Gen gewählt, dessen Gen-Knockout auf nur einem von beiden Allelen nicht letal ist, bei einem vollständigen Knockout (homozygot negativ) aber letal ist.[1] Weiterhin wird ein Meiose-spezifischer Promotor verwendet, der die Genexpression der Endonuklease in der Keimbahn verstärkt.[1] Das Gene-Drive-System breitet sich von einem Allel auf das Zweite aus. Aufgrund des Letalfaktors in der Keimbahn entsteht eine Unfruchtbarkeit, wenn beide Allele das Gene-Drive-System tragen (Homozygotie).
Zusätzliche Gene
Daneben können auch weitere Gene in das Gene-Drive-System eingefügt werden (Cargo-Gene). Beispielsweise können Resistenzfaktoren gegen Parasiten oder Viren in eine Population von Insekten eingeführt werden.[5][6][7][8] Das Gene-Drive-System kann verwendet werden, um die Geschlechtsanteile (männlich/weiblich) in einer Population zu verändern.[9]
Gene-Drive-Inaktivierung
Durch Verwendung eines weiteren Gene-Drive-Systems (englisch Cas9-triggered chain ablation, kurz CATCHA, basierend auf Cas9) könnte ein Gene-Drive-System in einer Population inaktiviert werden.[10]
2020 berichteten Forscher die Entwicklung von zwei Systemen für ein potenzielles Stoppen von Gene Drives, welche mittels CRISPR-Cas9 Genome Editing in Populationen in der freien Wildbahn eingeführt wurden. Ein Seniorautor des Artikels weist darauf hin, dass die zwei von den Wissenschaftlern laut der Studie getesteten Systeme kein falsches Gefühl der Sicherheit für den Einsatz von Gene Drives geben sollen.[11][12]
Sicherheit
Aufgrund der Ausbreitung von Gene-Drive-Systemen innerhalb einer Art unterliegen der Umgang und die Herstellung in Deutschland der biologischen Sicherheitsstufe 3. Ein Problem beim Gene Drive ist die Aufrechterhaltung eines Selektionsdrucks auf das Cargo-Gen (z. B. ein Resistenzfaktor), wenn dieser dem Wirt keinen Selektionsvorteil bringt, denn ein Gene-Drive-System mit einem mutierten und inaktivierten oder gar entfernten Cargo-Gen würde sich in Konkurrenz in der Population ausbreiten und das ursprüngliche verdrängen.[7] In einer 2018 veröffentlichten Studie wurde berichtet, dieses Problem gelöst zu haben. So konnte im Labor eine Mückenpopulation der Art Anopheles gambiae vollständig ausgerottet werden.[13]
Ein anderes Problem ist eine mögliche unbeabsichtigte Freisetzung, wodurch ein Gene-Drive-System sich in Populationen in der Wildnis ausbreiten würde.[14] Der Übergang auf eine Population in der Wildnis kann durch Verwendung einer Erkennungssequenz vermieden werden, die nur im Laborstamm vorkommt und sonst aber nicht in der Art. Bei induzierbaren Promotoren ohne Basalexpression wird in Abwesenheit des Induktors das Gene-Drive-System wie andere DNA ohne Gene Drive vererbt.[15]
Im August 2016 publizierte eine Gruppe von Umweltorganisationen, die sich mit synthetischer Biologie beschäftigen, die Civil Society Working Group on Gene Drives (Zivilgesellschaftliche Arbeitsgemeinschaft zu Gene-Drives) das Manifest Reckless Driving: Gene drives and the end of nature (Rücksichtsloser Antrieb: Gen-Drives und das Ende der Natur), um vor den ökologischen und gesellschaftlichen Folgen der Technik zu warnen.[16]
Vorsorgeprinzip
Mitte Dezember 2016 beschloss die 13. „Conference of the Parties to the Convention on Biological Diversity“ (COP CBD, Vertragsstaatenkonferenz bzw. „UN-Artenschutzgipfel“) in Cancún (Mexiko) die Anwendung des Vorsorgeprinzips auch auf Gene-Drives.[17] Im Juli 2020 wurde der Abschlussbericht des vom Bundesministerium für Bildung und Forschung geförderten, deutsch-österreichischen Forschungsprojekts GeneTip zu Risikoabschätzung im Sinne des Vorsorgeprinzips bei Gene Drives veröffentlicht.[18] Vor Beginn dieser Konferenz forderten 170 Gruppen angesichts der Gefahren ein Moratorium.[19]
Anwendungen
Gene-Drive-Systeme wurden in Hefen, Stechmücken und Fruchtfliegen angewendet.[20] Mögliche Anwendungen liegen in der Gentherapie.[20]
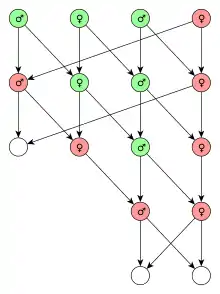
Die Bill & Melinda Gates Foundation versucht eine auf der Gen-Editiertechnik CRISPR/Cas9 basierende Methode zu entwickeln, den Überträger von Malaria, die Anopheles-Mücke, mit Hilfe des Gene-Drive-Prinzips auszurotten, um so die Malaria zu bekämpfen.[6] Stark vereinfacht funktioniert der Mechanismus zur Ausrottung der Art dadurch, dass alle Nachkommen einer manipulierten Mücke Träger eines rezessiven Gens sind, das aber erst dann zu einer Beeinträchtigung (zum Beispiel Unfruchtbarkeit oder nur männliche Nachkommen) führt, wenn Vater und Mutter Träger des Gens sind. Dass beide Elternteile das schädliche Gen tragen, wird durch den Gene Drive erreicht, der die Wahrscheinlichkeit, das schädliche Gen zu vererben, deutlich erhöht.
Lobbyarbeit
Eine Informationsfreiheitsanfrage zeigte,[21] dass die Bill & Melinda Gates Foundation[19] die Firma Emerging Ag bezahlte, um Lobbyarbeit gegen eine gesetzliche Regelung zu leisten. Als Zusammenschluss in der „Gene Drive Research Sponsors and Supporters coalition“ arbeitete man mit 65 Wissenschaftlern zusammen, die dem Forschungs- und Anwendungsbereich des Gene Drives im Rahmen des „UN CBD Online Forum“ und anderen Expertengremien Vorschub leisten sollten.
Bereits 2016 und 2017 produzierte die JASON Defense Advisory Group Berichte über die Technologie,[19] die zur biologischen Kriegsführung geeignet sein könnte. Forschung zu militärischen Zwecken finanziert in den USA die DARPA, mit US$ 100 Millionen bis 2017 sowie in Australien CSIRO.[19]
Literatur
- Gene Drives – Potenziale, Risiken, Besorgnisgründe. Ergebnisbericht des Pilot-Projekts 'GeneTip - Genetische Innovationen als Auslöser für Phasenübergänge in Populationsdynamiken von Tieren und Pflanzen‘ (PDF; 3,7 MB)
- Y. S. Chan, D. S. Huen, R. Glauert, E. Whiteway, S. Russell: Optimising homing endonuclease gene drive performance in a semi-refractory species: the Drosophila melanogaster experience. In: PloS one. Band 8, Nummer 1, 2013, S. e54130, doi:10.1371/journal.pone.0054130, PMID 23349805, PMC 3548849 (freier Volltext).
- J. Pugh: Driven to extinction? The ethics of eradicating mosquitoes with gene-drive technologies. In: Journal of medical ethics. [elektronische Veröffentlichung vor dem Druck] April 2016, doi:10.1136/medethics-2016-103462, PMID 27118691
- A Deplazes-Zemp, U Grossniklaus, F Lefort, P Müller, J Romeis, A Rüegsegger, N Schoenenberger, E Spehn (2020) Gene Drives: Nutzen, Risiken und mögliche Anwendungen. Swiss Academies Factsheets 15 (4), DOI:10.5281/zenodo.3742775
Rezeption
- Marc Elsberg: Helix. Sie werden uns ersetzen. (Roman, 2016)[22]
Siehe auch
Weblinks
- Kathrin Gießelmann und Eva Richter-Kuhlmann: Gene Drive: Das Ende der Vererbungsregeln Deutsches Ärzteblatt 37/2018
- Katrin Zöfel: Mit Gentechnik gegen Malaria, DLF – Forschung aktuell, 16. Dezember 2015
- Michael Lange: Die Gen-Bombe – Kettenreaktion gegen Zika, Malaria und Co., Deutschlandfunk – Wissenschaft im Brennpunkt vom 28. August 2016
- Lennart Pyritz und Michael Lange: Mit Genedrive gegen unerwünschte Arten – Selbstsüchtige Gene, Deutschlandfunk – Wissenschaft im Brennpunkt vom 24. September 2017
- Forschung aktuell, 28. August 2016: Genetische Kettenreaktion gegen Stechmücken
- Gene Drive: Gentechnik jetzt auch an wildlebenden Arten?, Umweltinstitut München
- Synthetische Biologie erklärt, Akademie der Naturwissenschaften Schweiz (SCNAT)
Einzelnachweise
- A. Burt: Site-specific selfish genes as tools for the control and genetic engineering of natural populations. In: Proceedings. Biological sciences / The Royal Society. Band 270, Nummer 1518, Mai 2003, S. 921–928, doi:10.1098/rspb.2002.2319, PMID 12803906, PMC 1691325 (freier Volltext).
- A. K. Lindholm, K. A. Dyer, R. C. Firman, L. Fishman, W. Forstmeier, L. Holman, H. Johannesson, U. Knief, H. Kokko, A. M. Larracuente, A. Manser, C. Montchamp-Moreau, V. G. Petrosyan, A. Pomiankowski, D. C. Presgraves, L. D. Safronova, A. Sutter, R. L. Unckless, R. L. Verspoor, N. Wedell, G. S. Wilkinson, T. A. Price: The Ecology and Evolutionary Dynamics of Meiotic Drive. In: Trends in ecology & evolution. Band 31, Nummer 4, April 2016, S. 315–326, doi:10.1016/j.tree.2016.02.001, PMID 26920473.
- K. M. Esvelt, A. L. Smidler, F. Catteruccia, G. M. Church: Concerning RNA-guided gene drives for the alteration of wild populations. In: eLife. [elektronische Veröffentlichung vor dem Druck] Juli 2014, doi:10.7554/eLife.03401, PMID 25035423, PMC 4117217 (freier Volltext).
- V. M. Gantz, E. Bier: Genome editing. The mutagenic chain reaction: a method for converting heterozygous to homozygous mutations. In: Science. Band 348, Nummer 6233, April 2015, S. 442–444, doi:10.1126/science.aaa5945, PMID 25908821, PMC 4687737 (freier Volltext).
- N. Windbichler, M. Menichelli, P. A. Papathanos, S. B. Thyme, H. Li, U. Y. Ulge, B. T. Hovde, D. Baker, R. J. Monnat, A. Burt, A. Crisanti: A synthetic homing endonuclease-based gene drive system in the human malaria mosquito. In: Nature. Band 473, Nummer 7346, Mai 2011, S. 212–215, doi:10.1038/nature09937, PMID 21508956, PMC 3093433 (freier Volltext).
- A. Hammond, R. Galizi, K. Kyrou, A. Simoni, C. Siniscalchi, D. Katsanos, M. Gribble, D. Baker, E. Marois, S. Russell, A. Burt, N. Windbichler, A. Crisanti, T. Nolan: A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. In: Nature Biotechnology. Band 34, Nummer 1, Januar 2016, S. 78–83, doi:10.1038/nbt.3439, PMID 26641531. PMC 4913862 (freier Volltext).
- Y. S. Chan, D. A. Naujoks, D. S. Huen, S. Russell: Insect population control by homing endonuclease-based gene drive: an evaluation in Drosophila melanogaster. In: Genetics. Band 188, Nummer 1, Mai 2011, S. 33–44, doi:10.1534/genetics.111.127506, PMID 21368273, PMC 3120159 (freier Volltext).
- V. M. Gantz, N. Jasinskiene, O. Tatarenkova, A. Fazekas, V. M. Macias, E. Bier, A. A. James: Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. In: Proceedings of the National Academy of Sciences. Band 112, Nummer 49, Dezember 2015, S. E6736–E6743, doi:10.1073/pnas.1521077112, PMID 26598698, PMC 4679060 (freier Volltext).
- Z. N. Adelman, Z. Tu: Control of Mosquito-Borne Infectious Diseases: Sex and Gene Drive. In: Trends in parasitology. Band 32, Nummer 3, März 2016, S. 219–229, doi:10.1016/j.pt.2015.12.003, PMID 26897660, PMC 4767671 (freier Volltext).
- B. Wu, L. Luo, X. J. Gao: Cas9-triggered chain ablation of cas9 as a gene drive brake. In: Nature Biotechnology. Band 34, Nummer 2, Februar 2016, S. 137–138, doi:10.1038/nbt.3444, PMID 26849513.
- Biologists create new genetic systems to neutralize gene drives In: phys.org. Abgerufen am 8. Oktober 2020 (englisch).
- Xiang-Ru Shannon Xu, Emily A. Bulger, Valentino M. Gantz, Carissa Klanseck, Stephanie R. Heimler, Ankush Auradkar, Jared B. Bennett, Lauren Ashley Miller, Sarah Leahy, Sara Sanz Juste, Anna Buchman, Omar S. Akbari, John M. Marshall, Ethan Bier: Active Genetic Neutralizing Elements for Halting or Deleting Gene Drives. In: Molecular Cell. 18. September 2020, ISSN 1097-2765, doi:10.1016/j.molcel.2020.09.003 (englisch, sciencedirect.com [abgerufen am 8. Oktober 2020]).
- Kyros Kyrou, Andrew M Hammond, Roberto Galizi, Nace Kranjc, Austin Burt, Andrea K Beaghton, Tony Nolan, Andrea Crisanti: A CRISPR–Cas9 gene drive targeting doublesex causes complete population suppression in caged Anopheles gambiae mosquitoes. In: Nature Biotechnology. 2018, doi:10.1038/nbt.4245.
- O. S. Akbari, H. J. Bellen, E. Bier, S. L. Bullock, A. Burt, G. M. Church, K. R. Cook, P. Duchek, O. R. Edwards, K. M. Esvelt, V. M. Gantz, K. G. Golic, S. J. Gratz, M. M. Harrison, K. R. Hayes, A. A. James, T. C. Kaufman, J. Knoblich, H. S. Malik, K. A. Matthews, K. M. O'Connor-Giles, A. L. Parks, N. Perrimon, F. Port, S. Russell, R. Ueda, J. Wildonger: BIOSAFETY. Safeguarding gene drive experiments in the laboratory. In: Science. Band 349, Nummer 6251, August 2015, S. 927–929, doi:10.1126/science.aac7932, PMID 26229113, PMC 4692367 (freier Volltext).
- ZKBS: Az. 45310.0111, Februar 2016: Stellungnahme der ZKBS zur Einstufung von gentechnischen Arbeiten zur Herstellung und Verwendung von höheren Organismen mit rekombinanten Gene-Drive-Systemen. Abgerufen am 28. Februar 2016.
- synbiowatch.org: Reckless Driving: Gene drives and the end of nature (2. September 2016)
- badische-zeitung.de, Wirtschaft, 20. Dezember 2016, Christian Mihatsch: UN-Konferenz stellt Weichen für Erhalt der Tier- und Pflanzenvielfalt (20. Dezember 2016)
- BioTip-Pilotstudie, auf genetip.de
- Heinrich-Böll-Stiftung Die Gene Drive Files (2017-12-05)
- V. M. Gantz, E. Bier: The dawn of active genetics. In: BioEssays : news and reviews in molecular, cellular and developmental biology. Band 38, Nummer 1, Januar 2016, S. 50–63, doi:10.1002/bies.201500102, PMID 26660392.
- Freigegebene Dokumente, genannt Gene Drive Files
- deutschlandfunk.de, Forschung aktuell, 1. November 2016, Ralf Krauter: Gene im Dienste der Dramaturgie (20. Dezember 2016)